Fitopatometria
Diretório das imagens
# mudar de acordo com a pasta em seu computador
setwd("E:/Desktop/tiagoolivoto/static/tutorials/pliman_esalq/leaves")
Severidade da doença
Utilizando paletas
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
library(pliman)
## |=======================================================|
## | Tools for Plant Image Analysis (pliman 0.3.0) |
## | Author: Tiago Olivoto |
## | Type 'vignette('pliman_start')' for a short tutorial |
## | Visit 'https://bit.ly/pliman' for a complete tutorial |
## |=======================================================|
img <- image_import("img_1.jpeg")
h <- image_import("h_img1.png")
d <- image_import("d_img1.png")
b <- image_import("b_img1.png")
image_combine(img, h, d, b, ncol = 4)

Padrão da função
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE)

## healthy symptomatic
## 76.32428 23.67572
Mostrando preenchimento das lesões
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
show_contour = FALSE)

## healthy symptomatic
## 75.52765 24.47235
Mostrando uma máscara
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
show_contour = FALSE,
show_original = FALSE)

## healthy symptomatic
## 75.71474 24.28526
Segmentando as lesões
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
show_contour = FALSE, # não mostra os contornos
segment = TRUE, # segmenta as lesões que se tocam por poucos pixeis
show_segmentation = TRUE) # mostra as segmentações

## healthy symptomatic
## 76.66014 23.33986
Analisar as lesões
feat <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
segment = TRUE,
show_features = TRUE, # computa características das lesões
lesion_size = "medium") # padrão

## healthy symptomatic
## 76.08522 23.91478
print_tbl(feat$statistics)
| stat | value |
|---|---|
| n | 424.000 |
| min_area | 35.000 |
| mean_area | 429.205 |
| max_area | 6644.000 |
| sd_area | 577.445 |
| sum_area | 181983.000 |
print_tbl(feat$shape[1:10, ])
| id | x | y | area | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | major_axis | eccentricity | theta |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 1060.298 | 274.790 | 6644 | 399 | 46.650 | 19.960 | 71.955 | 11.549 | 3.605 | 118.041 | 0.683 | -0.047 |
| 2 | 1058.626 | 361.842 | 5354 | 362 | 40.750 | 25.442 | 53.924 | 6.355 | 2.119 | 89.904 | 0.420 | -1.512 |
| 3 | 463.372 | 536.354 | 6297 | 432 | 45.609 | 27.939 | 61.951 | 8.971 | 2.217 | 115.499 | 0.765 | -0.083 |
| 4 | 635.068 | 690.510 | 2892 | 290 | 30.920 | 15.833 | 45.253 | 6.987 | 2.858 | 73.518 | 0.600 | 1.256 |
| 5 | 773.982 | 402.086 | 1768 | 163 | 23.417 | 18.567 | 30.553 | 2.941 | 1.646 | 54.829 | 0.637 | 1.081 |
| 6 | 800.030 | 317.214 | 2856 | 271 | 30.672 | 14.397 | 43.828 | 7.355 | 3.044 | 82.335 | 0.799 | 0.738 |
| 7 | 1123.307 | 352.000 | 2004 | 211 | 25.378 | 15.157 | 35.924 | 4.673 | 2.370 | 61.036 | 0.668 | -0.601 |
| 8 | 331.393 | 535.152 | 1187 | 140 | 19.208 | 12.609 | 24.671 | 2.776 | 1.957 | 45.128 | 0.626 | -1.537 |
| 9 | 1109.321 | 496.102 | 957 | 114 | 17.169 | 13.063 | 22.222 | 2.110 | 1.701 | 38.183 | 0.479 | -0.586 |
| 10 | 1091.068 | 470.443 | 1299 | 154 | 20.077 | 11.530 | 26.759 | 3.733 | 2.321 | 47.547 | 0.577 | -1.130 |
# corrigir as medidas (dpi = 150)
feat_corrected <- get_measures(feat, dpi = 150)
feat <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
segment = TRUE,
show_features = TRUE, # computa características das lesões
lesion_size = "large")

## healthy symptomatic
## 75.93441 24.06559
O valor de tolerance define a tolerancia para segmentação de objetos que se encostam. Menores valores tendem a dividir objetos mais facilmente.
feat <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_image = TRUE,
segment = TRUE,
tolerance = 0.2)

## healthy symptomatic
## 76.12413 23.87587
A little bit more!
# library(pliman)
sev_img2 <-
measure_disease(img = "img_2",
img_healthy = "h_img2",
img_symptoms = "d_img2",
img_background = "b_img2",
show_image = FALSE,
show_contour = FALSE,
col_background = "black")
## healthy symptomatic
## 91.58738 8.412622
imgs <- image_import(c("img_2.jpeg", "mask_im2.jpeg"))
image_combine(imgs)

Mais de uma folha
sev_folhas <-
measure_disease(img = "soy_21",
img_healthy = "h_s",
img_symptoms = "d_s",
img_background = "b_s",
show_image = TRUE,
save_image = TRUE,
show_contour = FALSE,
col_background = "black")
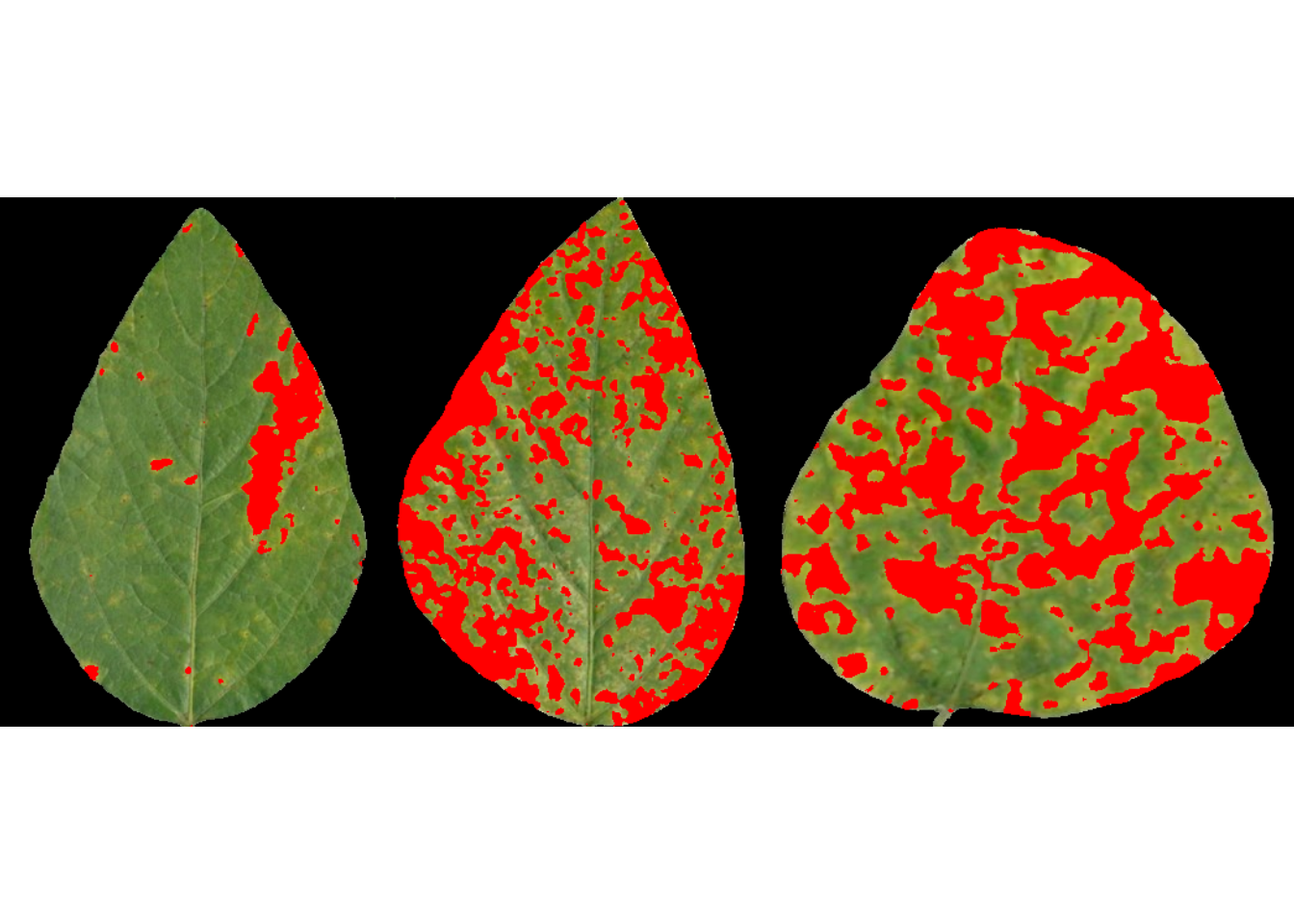
## healthy symptomatic
## 70.15104 29.84896
Utilizando índices RGB
Para identificar o melhor índice para segmentar a imagem do fundo e após as lesões da folha sadia, pode-se utilizar a função image_segment_iter. (OBS. somente funcionará em uma seção iterativa).
image_segment_iter(img)
Após escolhidos os índices, podemos utilizar os argumentos index_lb e index_dh para segmentação da folha e fundo | lesão e sadio, respectivamente.
# após escolhidos os índices, utiliza
sev_index <-
measure_disease(img,
index_lb = "G",
index_dh = "NGRDI",
show_image = TRUE)

## healthy symptomatic
## 74.34505 25.65495
Processamento em lote
Para analisar diversas imagens de um diretório, utiliza-se o argumento pattern, para declarar um padrão de nomes de arquivos. Serão utilizadas 15 folhas de soja disponíveis no repositório https://osf.io/4hbr6, um banco de dados de imagens de anotação de severidade de doenças de plantas. Obrigado a
Emerson M. Del Ponte e seus colaboradores por manter este projeto disponível publicamente.
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "h_s",
img_symptoms = "d_s",
img_background = "b_s",
verbose = FALSE)
)
## usuário sistema decorrido
## 15.05 0.97 16.14
print_tbl(sev_lote$severity)
| img | healthy | symptomatic |
|---|---|---|
| proc_soy_21 | 56.096 | 43.904 |
| soy_1 | 92.473 | 7.527 |
| soy_10 | 84.729 | 15.271 |
| soy_11 | 15.779 | 84.221 |
| soy_12 | 64.419 | 35.581 |
| soy_13 | 78.416 | 21.584 |
| soy_14 | 61.373 | 38.627 |
| soy_15 | 60.489 | 39.511 |
| soy_16 | 41.573 | 58.427 |
| soy_17 | 78.194 | 21.806 |
| soy_18 | 54.525 | 45.475 |
| soy_19 | 87.727 | 12.273 |
| soy_2 | 62.120 | 37.880 |
| soy_20 | 51.608 | 48.392 |
| soy_21 | 70.733 | 29.267 |
| soy_3 | 59.113 | 40.887 |
| soy_4 | 44.320 | 55.680 |
| soy_5 | 83.497 | 16.503 |
| soy_6 | 73.264 | 26.736 |
| soy_7 | 76.791 | 23.209 |
| soy_8 | 70.425 | 29.575 |
| soy_9 | 57.430 | 42.570 |
# exporta os resultados
# rio::export(sev_lote$severity, "severidade.xlsx")
Para acelerar o tempo de processamento quando várias imagens estão disponíveis, pode-se utilizar o argumento paralell. Isto criará múltiplas seções R em segundo plano, sendo cada uma responsável pelo processamento de uma parte das imagens.
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "h_s",
img_symptoms = "d_s",
img_background = "b_s",
verbose = FALSE,
parallel = TRUE)
)
## usuário sistema decorrido
## 0.22 0.03 10.17
Desafios
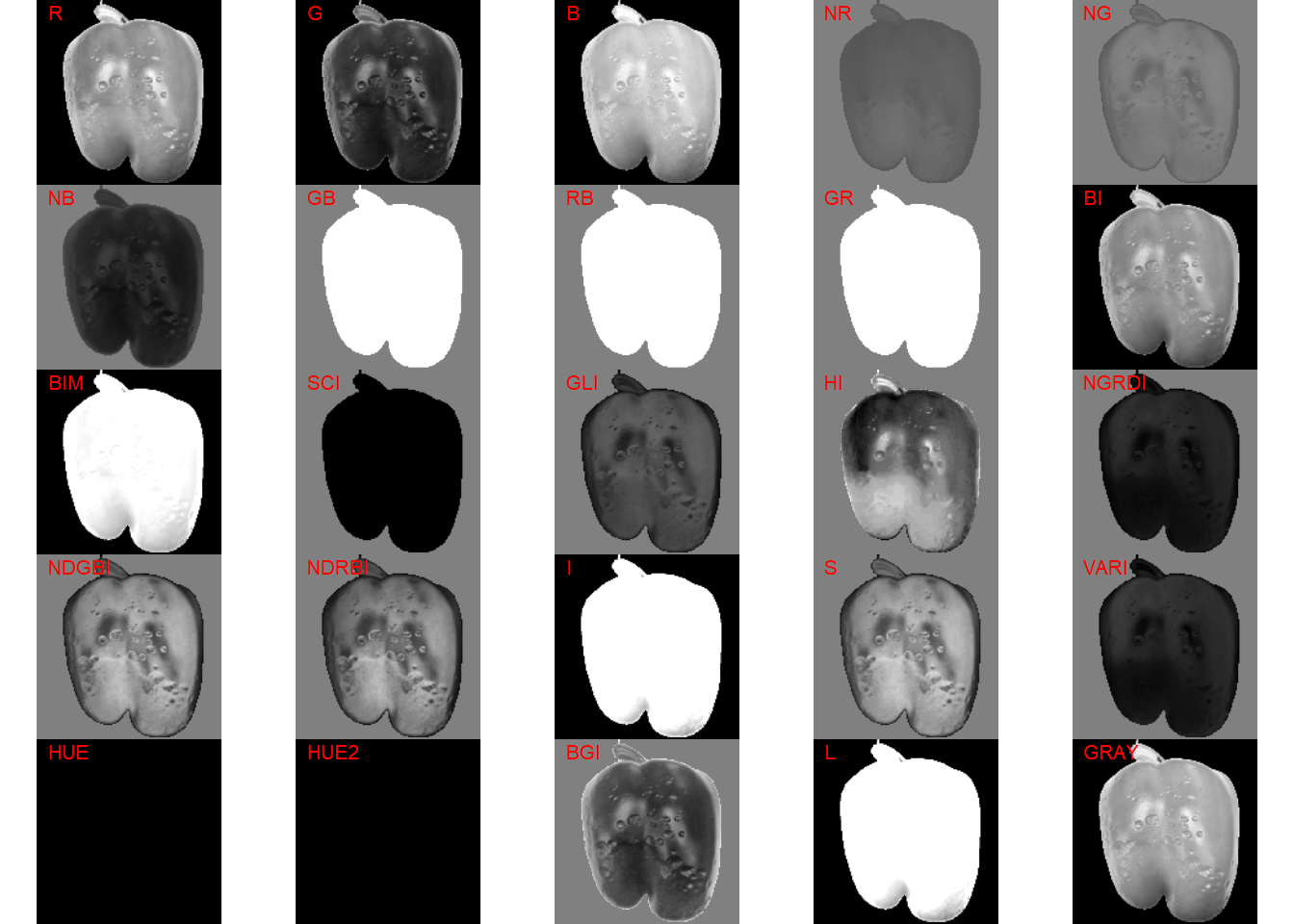
Lesões com pouco contraste
pepper <- image_import("pepper.png", plot = TRUE)

image_index(pepper, index = "all")
Fundos complexos
img <- image_import("maize_1.png", plot = TRUE)

image_segment(img, index = "all")

img <- image_import("maize_2.png", plot = TRUE)

image_segment(img, index = "all")

img2 <- image_crop(img,
width = 959:32,
height = 163:557,
plot = TRUE)
image_segment_iter(img2,
nseg = 2, # define o número de segmentações
index = c("R/(G+B)", "GLI"), # índices para primeira e segunda
invert = c(T, F), # inverter a segmentação? (passa um vetor)
ncol = 3) # número de colunas no plot
## image pixels percent
## 1 original 1466240 100.00000
## 2 seg1 72072 4.91543
## 3 seg2 17214 23.88445

img <- image_import("maize_3.png", plot = TRUE)

image_segment(img, index = "all")
