Analizando objetos
Diretório das imagens
# mudar de acordo com a pasta em seu computador
setwd("E:/Desktop/tiagoolivoto/static/tutorials/pliman_lca/imgs")
A função analyze_objects() pode ser usada para contar objetos em uma imagem. Vamos começar com um exemplo simples com a imagem object_300dpi.png disponível na
página GitHub. Para facilitar a importação de imagens desta pasta, uma função auxiliar image_pliman() é usada.
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
library(pliman)
## |==========================================================|
## | Tools for Plant Image Analysis (pliman 1.0.0) |
## | Author: Tiago Olivoto |
## | Type 'vignette('pliman_start')' for a short tutorial |
## | Visit 'http://bit.ly/pkg_pliman' for a complete tutorial |
## |==========================================================|
library(tidyverse)
## -- Attaching packages --------------------------------------- tidyverse 1.3.1 --
## v ggplot2 3.3.5 v purrr 0.3.4
## v tibble 3.1.6 v dplyr 1.0.7
## v tidyr 1.1.4 v stringr 1.4.0
## v readr 2.1.1 v forcats 0.5.1
## Warning: package 'tidyr' was built under R version 4.1.1
## Warning: package 'readr' was built under R version 4.1.2
## -- Conflicts ------------------------------------------ tidyverse_conflicts() --
## x forcats::%>%() masks stringr::%>%(), dplyr::%>%(), purrr::%>%(), tidyr::%>%(), tibble::%>%(), pliman::%>%()
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
library(patchwork)
img <- image_pliman("objects_300dpi.jpg", plot = TRUE)

A imagem acima foi produzida com o Microsoft PowerPoint. Tem uma resolução conhecida de 300 dpi(pontos por polegada) e mostra quatro objetos
- Quadrado maior: 10 x 10 cm (100 cm$^2$)
- Quadrado menor: 5 x 5 cm(25 cm$^2$)
- Retângulo: 4 x 2 cm(8 cm$^2$)
- Círculo: 3 cm de diâmetro(~7,07 cm$^2$)
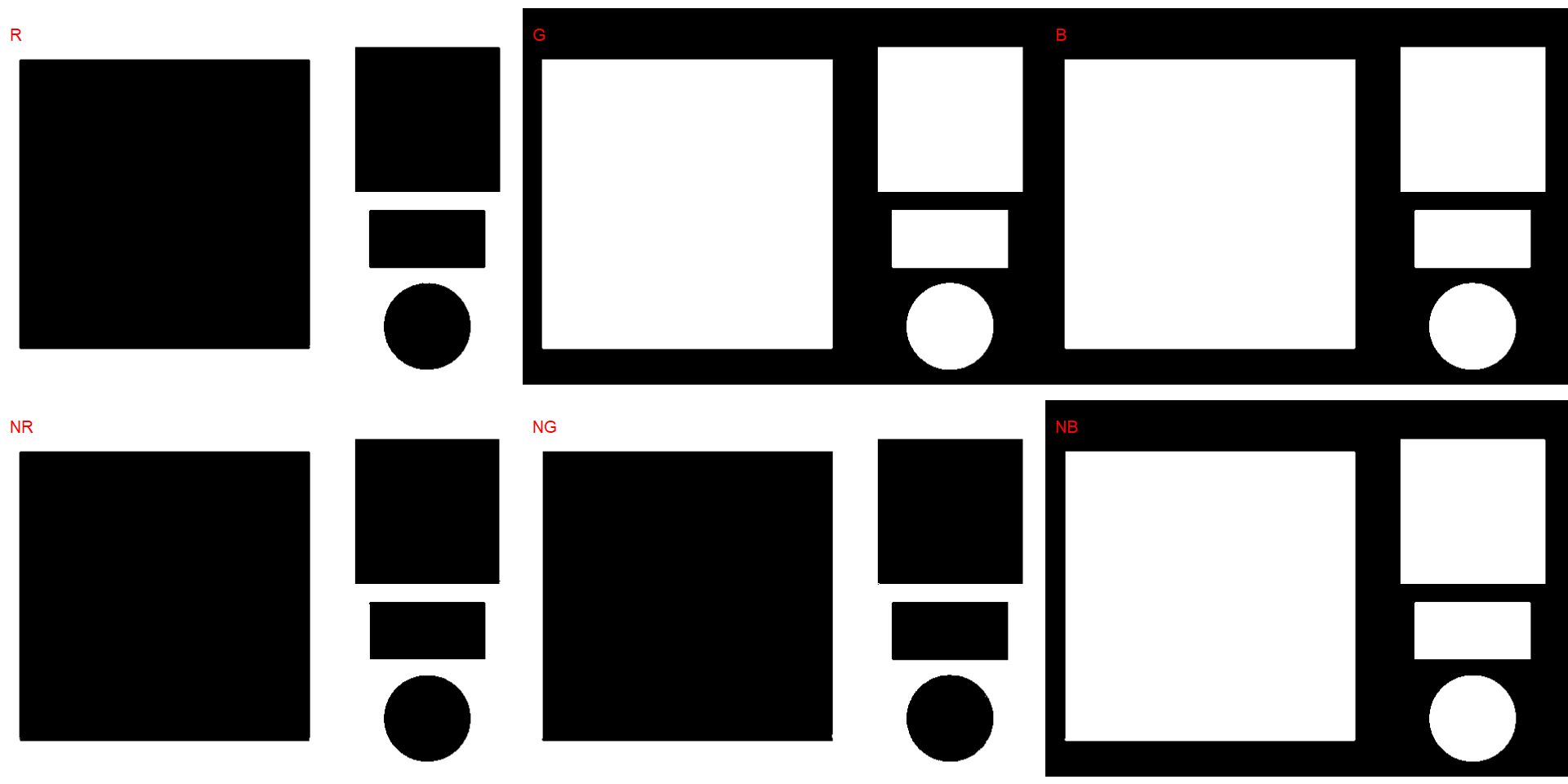
Para contar os objetos na imagem usamos analyze_objects() e informamos a imagem (o único argumento obrigatório). Primeiro, usamos image_binary() para ver o índice mais adequado para segmentar os objetos do fundo. Por padrão, R, G, B e seus valores normalizados.
image_binary(img)
Analizar objetos
img_res <- analyze_objects(img, marker = "id")

Ajustando as medidas do objeto
Os resultados foram armazenados em img_res. Como não há escala declarada no exemplo acima, não temos ideia sobre a área real dos objetos em cm$^2$, apenas em pixels. Neste caso, usamos get_measures() para ajustar as medidas de pixels para unidades métricas.
Existem duas formas principais de ajustar as medidas do objeto (de pixels a cm, por exemplo). O primeiro é declarar a área, perímetro ou raio conhecido de um determinado objeto. A medida para os outros objetos será então calculada por uma regra de três simples. A segunda é declarando uma resolução de imagem conhecida em dpi (pontos por polegada). Neste caso, perímetro, área e raio serão ajustados pelo dpi informado.
Declarando um valor conhecido
Como conhecemos a área do quadrado maior (objeto 1), vamos ajustar a área dos outros objetos na imagem usando isso.
get_measures(img_res,
id = 1,
area ~ 100)
## -----------------------------------------
## measures corrected with:
## object id: 1
## area : 100
## -----------------------------------------
## Total : 139.985
## Average : 34.996
## -----------------------------------------
## id x y area area_ch perimeter radius_mean radius_min
## 1 1 668.506 798.002 100.000 99.915 39.932 5.733 4.992
## 2 2 1737.513 453.246 25.041 24.978 19.924 2.865 2.495
## 3 3 1737.575 1296.331 7.023 7.015 8.504 1.491 1.483
## 4 4 1737.972 939.503 7.922 7.880 11.886 1.666 0.986
## radius_max radius_sd radius_ratio diam_mean diam_min diam_max major_axis
## 1 7.062 74.203 1.415 11.466 9.983 14.124 11.552
## 2 3.524 36.957 1.413 5.730 4.989 7.049 5.781
## 3 1.502 0.398 1.012 2.983 2.967 3.003 2.991
## 4 2.222 50.007 2.253 3.332 1.973 4.444 4.611
## minor_axis eccentricity theta solidity circularity
## 1 11.542 0.041 0.014 1.001 0.788
## 2 5.776 0.041 1.539 1.003 0.793
## 3 2.990 0.026 -1.490 1.001 1.220
## 4 2.291 0.868 0.000 1.005 0.705
O mesmo pode ser usado para ajustar as medidas com base no perímetro ou raio. Vamos ajustar o perímetro dos objetos pelo perímetro do objeto 2(20 cm).
Declarando a resolução da imagem
Se a resolução da imagem for conhecida, todas as medidas serão ajustadas de acordo com esta resolução. Vamos ver um exemplo numérico com pixels_to_cm(). Esta função converte o número de pixels (px) em cm, considerando a resolução da imagem em dpi, da seguinte forma: \(cm = px \times(2,54 / dpi)\). Como sabemos o número de pixels do quadrado maior, seu perímetro em cm é dado por
# número de pixels para o perímetro do quadrado maior
ls_px <- img_res$results$perimeter[1]
pixels_to_cm(px = ls_px, dpi = 300)
## [1] 39.878
O perímetro do objeto 1 ajustado pela resolução da imagem é muito próximo do verdadeiro (40 cm). Abaixo, os valores de todas as medidas são ajustados declarando o argumento dpi em get_measures().
img_res_cor <- get_measures(img_res, dpi = 300)
print_tbl(t(img_res_cor))
| 1 | 2 | 3 | 4 | |
|---|---|---|---|---|
| id | 1.000 | 2.000 | 3.000 | 4.000 |
| x | 668.506 | 1737.513 | 1737.575 | 1737.972 |
| y | 798.002 | 453.246 | 1296.331 | 939.503 |
| area | 99.729 | 24.973 | 7.004 | 7.900 |
| area_ch | 99.644 | 24.910 | 6.996 | 7.858 |
| perimeter | 39.878 | 19.897 | 8.492 | 11.870 |
| radius_mean | 5.725 | 2.861 | 1.489 | 1.664 |
| radius_min | 4.985 | 2.491 | 1.481 | 0.985 |
| radius_max | 7.052 | 3.520 | 1.500 | 2.219 |
| radius_sd | 74.203 | 36.957 | 0.398 | 50.007 |
| radius_ratio | 1.415 | 1.413 | 1.012 | 2.253 |
| diam_mean | 11.450 | 5.722 | 2.979 | 3.327 |
| diam_min | 9.970 | 4.983 | 2.963 | 1.970 |
| diam_max | 14.105 | 7.039 | 2.999 | 4.438 |
| major_axis | 11.536 | 5.773 | 2.987 | 4.604 |
| minor_axis | 11.526 | 5.768 | 2.986 | 2.288 |
| eccentricity | 0.041 | 0.041 | 0.026 | 0.868 |
| theta | 0.014 | 1.539 | -1.490 | 0.000 |
| solidity | 1.001 | 1.003 | 1.001 | 1.005 |
| circularity | 0.788 | 0.793 | 1.220 | 0.705 |
Entendendo as medidas
object_contour(img) %>% # obtém o contorno dos objetos
poly_mass() %>% # computa o centro de massa e raios mínimo e máximo
plot_mass() # plota as medidas
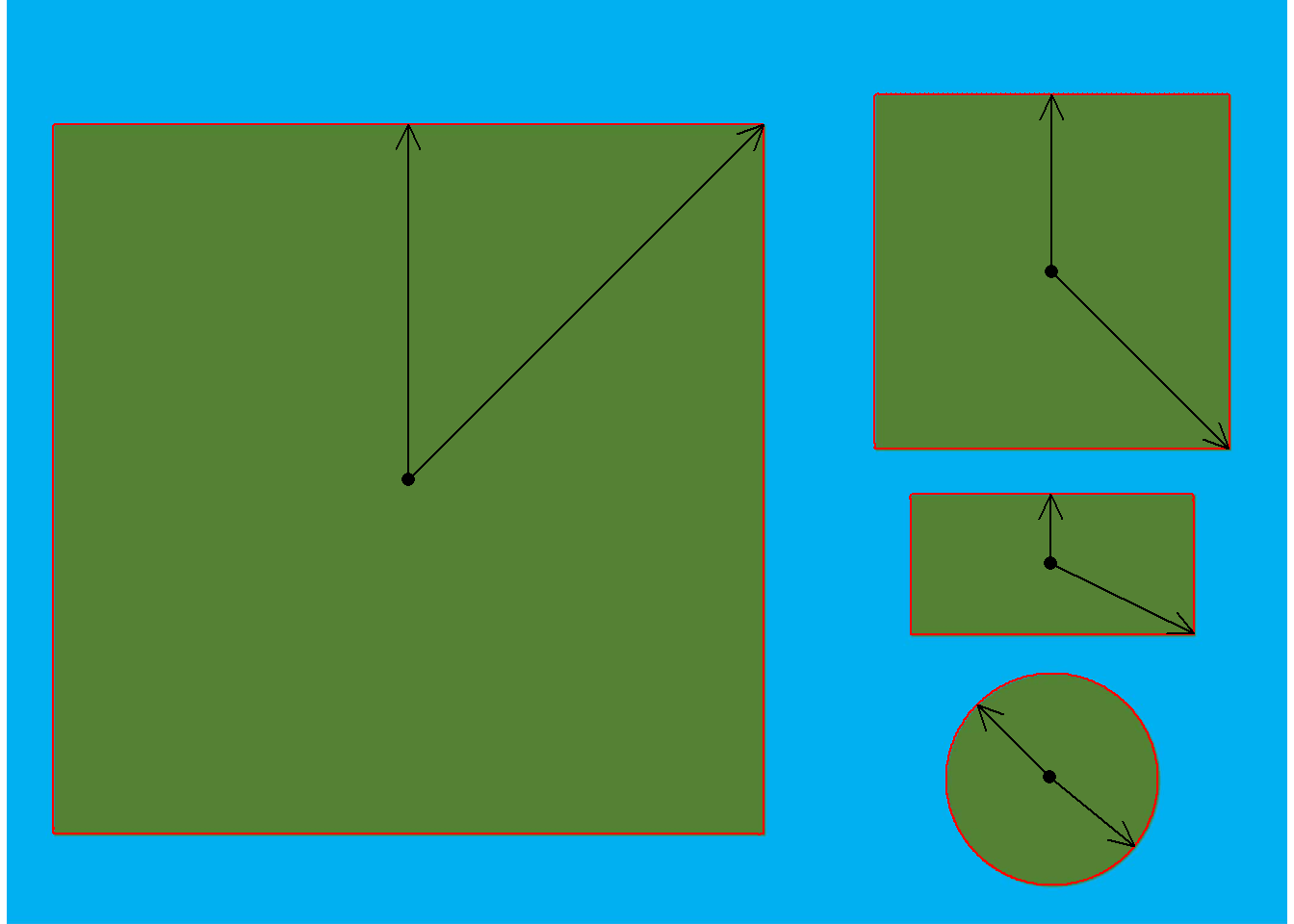
- Quadrado maior:
-
O diâmetro mínimo (a = 9,97) pode ser considerado como o lado do quadrado
-
O diâmetro máximo, dado por
\(a \sqrt{2}\)pode ser considerado a diagonal do quadrado ($9,97 \sqrt{2} = 14.099$
t(img_res_cor)
## 1 2 3 4
## id 1.000 2.000 3.000 4.000
## x 668.506 1737.513 1737.575 1737.972
## y 798.002 453.246 1296.331 939.503
## area 99.729 24.973 7.004 7.900
## area_ch 99.644 24.910 6.996 7.858
## perimeter 39.878 19.897 8.492 11.870
## radius_mean 5.725 2.861 1.489 1.664
## radius_min 4.985 2.491 1.481 0.985
## radius_max 7.052 3.520 1.500 2.219
## radius_sd 74.203 36.957 0.398 50.007
## radius_ratio 1.415 1.413 1.012 2.253
## diam_mean 11.450 5.722 2.979 3.327
## diam_min 9.970 4.983 2.963 1.970
## diam_max 14.105 7.039 2.999 4.438
## major_axis 11.536 5.773 2.987 4.604
## minor_axis 11.526 5.768 2.986 2.288
## eccentricity 0.041 0.041 0.026 0.868
## theta 0.014 1.539 -1.490 0.000
## solidity 1.001 1.003 1.001 1.005
## circularity 0.788 0.793 1.220 0.705
Objetos médios
Aqui, contaremos os grãos na imagem soybean_touch.jpg. Esta imagem tem um fundo ciano e contém 30 grãos de soja que se tocam. A função analyze_objects() segmenta a imagem usando como padrão o índice azul normalizado, como segue \(NB =(B /(R + G + B))\), onde R, G e B são as faixas vermelha, verde e azul. Os objetos são contados e os objetos segmentados são coloridos com permutações aleatórias.
soy <- image_pliman("soybean_touch.jpg")
count <- analyze_objects(soy)

count$statistics %>%
print_tbl()
| stat | value |
|---|---|
| n | 30.000 |
| min_area | 1366.000 |
| mean_area | 2057.367 |
| max_area | 2445.000 |
| sd_area | 230.557 |
| sum_area | 61721.000 |
Os usuários podem definir show_contour = FALSE para remover a linha de contorno e identificar os objetos (neste exemplo, os grãos) usando os argumentos marker = "id". A cor do fundo também pode ser alterada com col_background.
count2 <-
analyze_objects(soy,
show_contour = FALSE,
marker = "id",
show_segmentation = FALSE,
col_background = "white") # padrão
# Obtenha as medidas do objeto
medidas <- get_measures(count)
medidas %>%
print_tbl()
| id | x | y | area | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 245.833 | 509.841 | 2286 | 2321 | 158 | 26.543 | 22.763 | 28.941 | 1.425 | 1.271 | 53.086 | 45.526 | 57.883 | 56.422 | 51.849 | 0.394 | -0.870 | 0.985 | 1.151 |
| 2 | 538.056 | 401.896 | 2299 | 2258 | 153 | 26.607 | 24.957 | 28.400 | 0.935 | 1.138 | 53.214 | 49.914 | 56.800 | 56.604 | 51.733 | 0.406 | -0.838 | 1.018 | 1.234 |
| 3 | 237.592 | 339.825 | 2312 | 2282 | 152 | 26.699 | 23.965 | 29.044 | 1.233 | 1.212 | 53.398 | 47.930 | 58.088 | 57.510 | 51.247 | 0.454 | -0.572 | 1.013 | 1.258 |
| 4 | 345.357 | 105.783 | 2445 | 2406 | 158 | 27.513 | 24.682 | 30.471 | 1.730 | 1.235 | 55.027 | 49.365 | 60.942 | 60.922 | 51.105 | 0.544 | -0.991 | 1.016 | 1.231 |
| 5 | 406.931 | 77.549 | 2302 | 2264 | 153 | 26.649 | 23.965 | 29.636 | 1.640 | 1.237 | 53.298 | 47.931 | 59.272 | 58.860 | 49.819 | 0.533 | 1.144 | 1.017 | 1.236 |
| 6 | 277.445 | 260.559 | 2163 | 2120 | 149 | 25.766 | 24.291 | 27.887 | 0.765 | 1.148 | 51.532 | 48.581 | 55.774 | 53.963 | 51.097 | 0.322 | -0.163 | 1.020 | 1.224 |
| 7 | 301.206 | 370.092 | 2217 | 2202 | 154 | 26.113 | 23.591 | 28.687 | 1.358 | 1.216 | 52.227 | 47.182 | 57.375 | 56.484 | 50.034 | 0.464 | -1.493 | 1.007 | 1.175 |
| 8 | 192.828 | 379.645 | 2207 | 2176 | 149 | 26.105 | 23.715 | 28.858 | 1.488 | 1.217 | 52.210 | 47.431 | 57.717 | 57.339 | 49.030 | 0.518 | 0.956 | 1.014 | 1.249 |
| 9 | 434.710 | 553.707 | 2174 | 2132 | 148 | 25.890 | 23.750 | 28.506 | 1.437 | 1.200 | 51.781 | 47.499 | 57.012 | 56.797 | 48.755 | 0.513 | 0.946 | 1.020 | 1.247 |
| 10 | 594.744 | 47.311 | 2219 | 2182 | 153 | 26.160 | 23.352 | 29.659 | 1.856 | 1.270 | 52.320 | 46.704 | 59.319 | 58.590 | 48.249 | 0.567 | -1.032 | 1.017 | 1.191 |
| 11 | 468.997 | 56.425 | 2315 | 2275 | 155 | 26.765 | 23.031 | 30.780 | 2.349 | 1.336 | 53.531 | 46.061 | 61.560 | 61.230 | 48.157 | 0.618 | 1.292 | 1.018 | 1.211 |
| 12 | 461.172 | 156.027 | 2175 | 2131 | 148 | 25.933 | 23.071 | 29.512 | 1.656 | 1.279 | 51.866 | 46.142 | 59.024 | 57.396 | 48.284 | 0.541 | 1.090 | 1.021 | 1.248 |
| 13 | 202.075 | 203.461 | 2188 | 2166 | 153 | 26.008 | 22.487 | 29.808 | 1.987 | 1.326 | 52.015 | 44.974 | 59.616 | 58.474 | 47.719 | 0.578 | -1.131 | 1.010 | 1.175 |
| 14 | 403.486 | 169.015 | 2035 | 1994 | 143 | 25.019 | 22.398 | 27.010 | 1.168 | 1.206 | 50.038 | 44.796 | 54.020 | 54.002 | 48.012 | 0.458 | -1.094 | 1.021 | 1.251 |
| 15 | 245.987 | 221.375 | 2117 | 2091 | 148 | 25.528 | 21.950 | 29.113 | 1.793 | 1.326 | 51.056 | 43.900 | 58.226 | 56.809 | 47.542 | 0.547 | -1.282 | 1.012 | 1.215 |
| 16 | 250.400 | 436.934 | 1964 | 1928 | 142 | 24.552 | 22.971 | 26.258 | 0.798 | 1.143 | 49.104 | 45.943 | 52.515 | 51.553 | 48.573 | 0.335 | -1.383 | 1.019 | 1.224 |
| 17 | 84.671 | 206.432 | 2183 | 2144 | 151 | 25.923 | 22.825 | 28.743 | 1.708 | 1.259 | 51.846 | 45.650 | 57.486 | 57.492 | 48.382 | 0.540 | 0.023 | 1.018 | 1.203 |
| 18 | 448.412 | 296.209 | 2068 | 2023 | 145 | 25.196 | 23.357 | 27.032 | 0.993 | 1.157 | 50.392 | 46.714 | 54.063 | 53.898 | 48.857 | 0.422 | -1.555 | 1.022 | 1.236 |
| 19 | 296.178 | 186.505 | 2056 | 2012 | 144 | 25.132 | 22.639 | 27.213 | 1.211 | 1.202 | 50.265 | 45.279 | 54.426 | 54.444 | 48.094 | 0.469 | 1.549 | 1.022 | 1.246 |
| 20 | 321.973 | 321.691 | 1978 | 1982 | 151 | 24.635 | 21.223 | 27.370 | 1.595 | 1.290 | 49.271 | 42.445 | 54.739 | 52.546 | 48.202 | 0.398 | 1.518 | 0.998 | 1.090 |
| 21 | 550.202 | 200.506 | 1939 | 1902 | 141 | 24.400 | 22.578 | 26.353 | 0.849 | 1.167 | 48.799 | 45.156 | 52.706 | 51.869 | 47.628 | 0.396 | 0.733 | 1.019 | 1.226 |
| 22 | 106.294 | 432.089 | 1922 | 1886 | 140 | 24.304 | 22.838 | 26.264 | 0.891 | 1.150 | 48.608 | 45.675 | 52.528 | 51.942 | 47.122 | 0.421 | 0.759 | 1.019 | 1.232 |
| 23 | 242.940 | 388.543 | 1926 | 1942 | 146 | 24.391 | 22.412 | 27.420 | 0.958 | 1.223 | 48.781 | 44.825 | 54.840 | 50.517 | 48.757 | 0.262 | -0.901 | 0.992 | 1.135 |
| 24 | 492.988 | 344.441 | 1891 | 1855 | 139 | 24.076 | 21.894 | 25.988 | 1.180 | 1.187 | 48.152 | 43.789 | 51.976 | 52.247 | 46.096 | 0.471 | 1.505 | 1.019 | 1.230 |
| 25 | 721.705 | 586.342 | 1915 | 1873 | 140 | 24.250 | 21.886 | 26.818 | 1.350 | 1.225 | 48.500 | 43.772 | 53.636 | 53.127 | 45.908 | 0.503 | 1.339 | 1.022 | 1.228 |
| 26 | 510.468 | 158.372 | 1787 | 1767 | 137 | 23.466 | 21.173 | 26.180 | 1.009 | 1.236 | 46.932 | 42.345 | 52.360 | 48.654 | 47.026 | 0.257 | -1.172 | 1.011 | 1.196 |
| 27 | 92.838 | 569.395 | 1743 | 1708 | 134 | 23.124 | 21.249 | 25.332 | 1.044 | 1.192 | 46.248 | 42.499 | 50.663 | 50.016 | 44.394 | 0.461 | 0.812 | 1.020 | 1.220 |
| 28 | 281.037 | 474.071 | 1819 | 1923 | 154 | 23.484 | 17.887 | 27.203 | 2.497 | 1.521 | 46.968 | 35.773 | 54.405 | 49.847 | 47.188 | 0.322 | 0.105 | 0.946 | 0.964 |
| 29 | 273.273 | 547.459 | 1710 | 1726 | 143 | 22.780 | 17.617 | 25.910 | 1.792 | 1.471 | 45.561 | 35.234 | 51.820 | 49.140 | 44.737 | 0.414 | -0.600 | 0.991 | 1.051 |
| 30 | 265.292 | 143.411 | 1366 | 1331 | 117 | 20.405 | 18.590 | 22.030 | 0.772 | 1.185 | 40.809 | 37.181 | 44.060 | 43.697 | 39.816 | 0.412 | -0.461 | 1.026 | 1.254 |
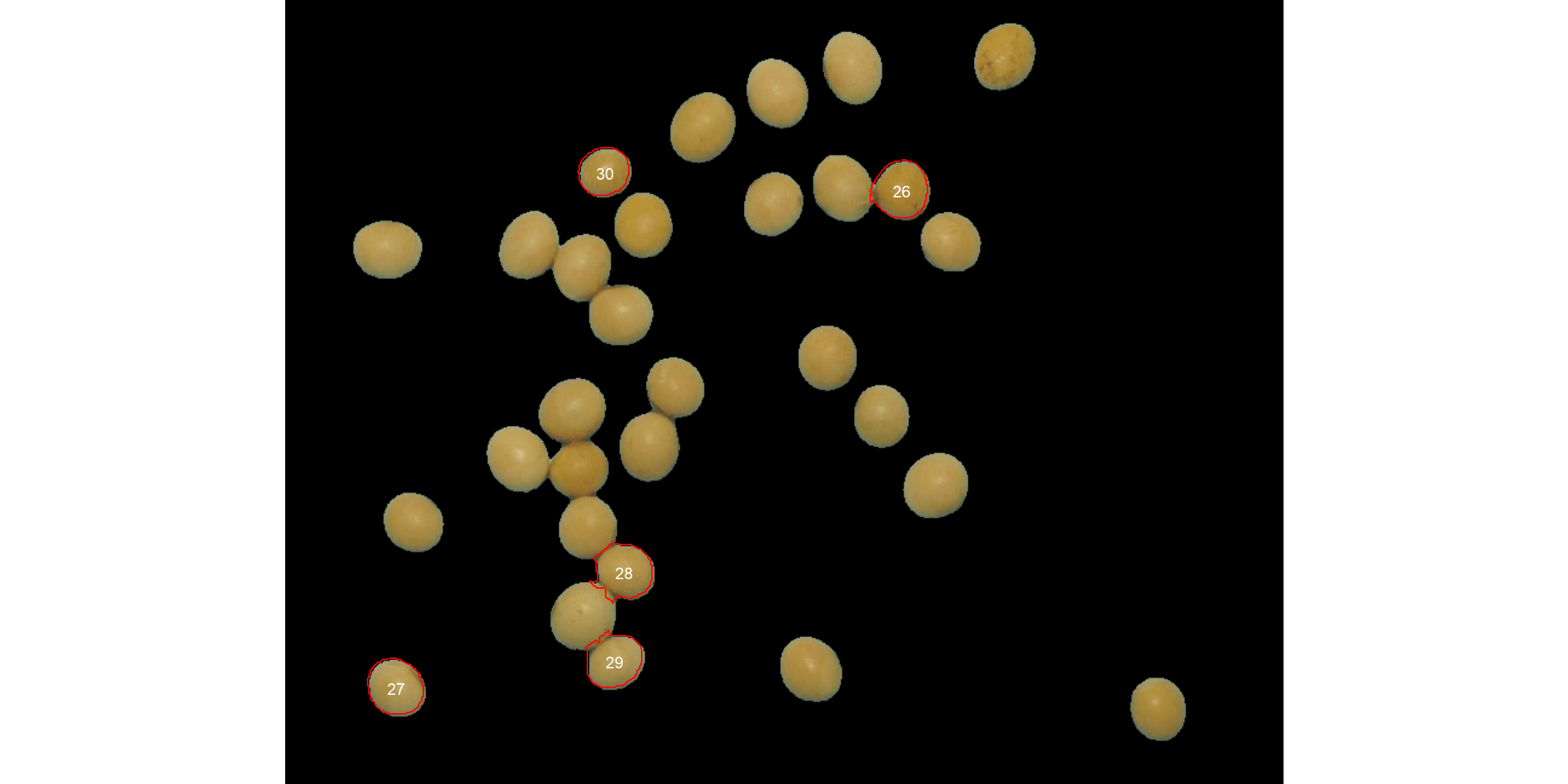
No exemplo a seguir, selecionaremos objetos com uma área acima da média de todos os objetos usando lower_size = 2057,36. Além disso, usaremos o argumento show_original = FALSE para mostrar os resultados como uma máscara.
analyze_objects(soy,
marker = "id",
show_original = FALSE,
lower_size = 2057.36)

Os usuários também podem usar os argumentos topn_* para selecionar os n objetos com base nas menores ou maiores áreas. Vamos ver como selecionar os 5 grãos com a menor área, mostrando os grãos originais em um fundo azul. Também usaremos o argumento my_index para escolher um índice personalizado para segmentar a imagem. Apenas para comparação, iremos configurar explicitamente o índice azul normalizado chamando my_index = "B/(R + G + B)".
analyze_objects(soy,
marker = "id",
topn_lower = 5,
col_background = "black",
my_index = "B /(R + G + B)")
Objetos grandes
cob <- image_import("cob.jpg")
# segmentação normal
analyze_objects(cob, marker = "id")

# ajustar o parâmetro object_size
cob_res <-
analyze_objects(cob,
object_size = "large",
marker = "id")

Objetos pequenos
wheat <- image_import("wheat.jpg")
analyze_objects(wheat)
vicia <- image_import("vicia2.jpg")
analyze_objects(vicia,
index = "R",
marker = "point",
marker_col = "red",
show_contour = FALSE)

Coordenadas de objetos
Os usuários podem obter as coordenadas para todos os objetos desejados com object_coord(). Quando o argumento id é definido como NULL (padrão), um retângulo delimitador é desenhado incluindo todos os objetos. Use id = "all" para obter as coordenadas de todos os objetos na imagem ou use um vetor numérico para indicar os objetos para calcular as coordenadas. Note que o argumento watershed = FALSE é usado para
folhas <- image_import(image = "leaves.jpg", plot = TRUE)
# obter o id de cada objeto
object_id(folhas, watershed = FALSE)

# Obtenha as coordenadas de um retângulo delimitador em torno de todos os objetos
object_coord(folhas, watershed = FALSE)

## $col_min
## [1] 47
##
## $col_max
## [1] 877
##
## $row_min
## [1] 35
##
## $row_max
## [1] 1112
# Obtenha as coordenadas para todos os objetos
object_coord(folhas,
id = "all",
watershed = FALSE)

## $col_min
## [1] 47 51 54 56 75 79 94 262 292 292 295 308 314 374 376 390 392 412 422
## [20] 463 482 499 526 529 537 540 544 597 603 610 642 661 684 703 709 710 710 714
## [39] 761 778 779
##
## $col_max
## [1] 52 372 294 411 308 88 261 686 296 569 308 510 483 378 380 394 529 598 426
## [20] 677 486 572 531 534 657 610 730 658 608 717 785 869 877 707 713 768 714 718
## [39] 765 796 783
##
## $row_min
## [1] 534 769 464 181 622 425 390 35 516 977 515 345 531 155 156 163 688 796 176
## [20] 450 556 622 360 359 325 699 916 783 782 555 720 152 802 720 403 318 722 723
## [39] 320 362 639
##
## $row_max
## [1] 538 1037 606 370 761 430 452 189 520 1112 520 422 662 159 160
## [16] 167 734 941 180 564 560 660 364 363 362 732 1019 896 786 658
## [31] 762 308 932 724 407 406 726 727 324 641 643
# Obtenha as coordenadas dos objetos 1 e 3
object_coord(folhas,
id = c(2, 4),
watershed = FALSE)

## $col_min
## [1] 51 56
##
## $col_max
## [1] 372 411
##
## $row_min
## [1] 769 181
##
## $row_max
## [1] 1037 370
Isolando objetos
Para isolar objetos, a função object_isolate() é usada. No exemplo a seguir, irei isolar o objeto 32 e definir uma borda de 5 pixels ao redor do objeto.
id1 <-
object_isolate(folhas,
watershed = FALSE,
id = 32,
edge = 5)
plot(id1)

Processamento em lote
Na análise de imagens, frequentemente é necessário processar mais de uma imagem. Por exemplo, no melhoramento de plantas, o número de grãos por planta (por exemplo, trigo) é frequentemente usado na seleção indireta de plantas de alto rendimento. No pliman, o processamento em lote pode ser feito quando o usuário declara o argumento pattern.
Para acelerar o tempo de processamento, especialmente para um grande número de imagens, o argumento parallel = TRUE pode ser usado. Nesse caso, as imagens são processadas de forma assíncrona(em paralelo) em sessões R separadas rodando em segundo plano na mesma máquina. O número de seções é configurado para 50% dos núcleos disponíveis. Este número pode ser controlado explicitamente com o argumento trabalhadores.
system.time(
list_res <- analyze_objects(pattern = "img_sb")
)
## Processing image img_sb_50_1 |=== | 8% 00:00:00

## Processing image img_sb_50_10 |===== | 15% 00:00:00

## Processing image img_sb_50_11 |======== | 23% 00:00:01

## Processing image img_sb_50_12 |========== | 31% 00:00:02

## Processing image img_sb_50_13 |============= | 38% 00:00:02

## Processing image img_sb_50_2 |================ | 46% 00:00:03

## Processing image img_sb_50_3 |=================== | 54% 00:00:04

## Processing image img_sb_50_4 |====================== | 62% 00:00:05

## Processing image img_sb_50_5 |======================== | 69% 00:00:06

## Processing image img_sb_50_6 |=========================== | 77% 00:00:07

## Processing image img_sb_50_7 |============================== | 85% 00:00:08

## Processing image img_sb_50_8 |================================ | 92% 00:00:09

## Processing image img_sb_50_9 |===================================| 100% 00:00:10
## --------------------------------------------
## Image Objects
## img_sb_50_1 100
## img_sb_50_10 29
## img_sb_50_11 23
## img_sb_50_12 15
## img_sb_50_13 7
## img_sb_50_2 90
## img_sb_50_3 83
## img_sb_50_4 75
## img_sb_50_5 70
## img_sb_50_6 60
## img_sb_50_7 57
## img_sb_50_8 48
## img_sb_50_9 36
## --------------------------------------------
## Done!

## usuário sistema decorrido
## 10.44 0.51 10.97
# procesamento paralelo
# três múltiplas seções
system.time(
list_res <-
analyze_objects(pattern = "img_sb",
show_image = FALSE,
parallel = TRUE,
workers = 3)
)
## Image processing using multiple sessions (3). Please wait.
## --------------------------------------------
## Image Objects
## img_sb_50_1 100
## img_sb_50_10 29
## img_sb_50_11 23
## img_sb_50_12 15
## img_sb_50_13 7
## img_sb_50_2 90
## img_sb_50_3 83
## img_sb_50_4 75
## img_sb_50_5 70
## img_sb_50_6 60
## img_sb_50_7 57
## img_sb_50_8 48
## img_sb_50_9 36
## --------------------------------------------
## Done!
## usuário sistema decorrido
## 0.03 0.03 3.01
list_res$count %>%
head() %>%
print_tbl()
| Image | Objects | |
|---|---|---|
| 1 | img_sb_50_1 | 100 |
| 7 | img_sb_50_10 | 29 |
| 13 | img_sb_50_11 | 23 |
| 19 | img_sb_50_12 | 15 |
| 25 | img_sb_50_13 | 7 |
| 31 | img_sb_50_2 | 90 |
list_res$results %>%
head() %>%
print_tbl()
| img | id | x | y | area | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| img_sb_50_1 | 1 | 518.854 | 260.189 | 185 | 171 | 44 | 7.197 | 5.645 | 8.276 | 0.644 | 1.466 | 14.393 | 11.289 | 16.553 | 16.777 | 14.083 | 0.543 | 0.874 | 1.082 | 1.201 |
| img_sb_50_1 | 2 | 468.818 | 398.552 | 192 | 175 | 43 | 7.357 | 6.306 | 8.202 | 0.415 | 1.301 | 14.715 | 12.612 | 16.404 | 16.288 | 15.046 | 0.383 | 1.105 | 1.097 | 1.305 |
| img_sb_50_1 | 3 | 332.133 | 442.092 | 173 | 160 | 42 | 6.969 | 6.169 | 7.854 | 0.370 | 1.273 | 13.938 | 12.338 | 15.707 | 15.059 | 14.694 | 0.219 | -0.425 | 1.081 | 1.232 |
| img_sb_50_1 | 4 | 242.217 | 269.533 | 184 | 168 | 42 | 7.230 | 6.163 | 8.288 | 0.540 | 1.345 | 14.460 | 12.326 | 16.577 | 16.748 | 13.979 | 0.551 | 1.074 | 1.095 | 1.311 |
| img_sb_50_1 | 5 | 365.399 | 453.936 | 188 | 171 | 42 | 7.310 | 6.243 | 8.210 | 0.481 | 1.315 | 14.621 | 12.485 | 16.419 | 16.734 | 14.297 | 0.520 | -0.659 | 1.099 | 1.339 |
| img_sb_50_1 | 6 | 540.210 | 199.448 | 181 | 167 | 44 | 7.138 | 6.252 | 8.087 | 0.463 | 1.294 | 14.276 | 12.504 | 16.175 | 15.695 | 14.813 | 0.330 | 0.806 | 1.084 | 1.175 |
Forma de objetos
A função analyze_objects() calcula uma gama de medidas que podem ser utilizadas para estudar a forma dos objetos, como por exemplo, folhas. Como exemplo, usarei a imagem potato_leaves.png, que foi coletada de
Gupta et al.(2020)
batata <- image_pliman("potato_leaves.jpg", plot = TRUE)
pot_meas <-
analyze_objects(batata,
watershed = FALSE,
marker = "id",
show_chull = TRUE) # mostra o casco convex

pot_meas$results %>%
print_tbl()
| id | x | y | area | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 854.542 | 224.043 | 51380 | 54536 | 852 | 131.565 | 92.111 | 198.025 | 26.061 | 2.150 | 263.131 | 184.222 | 396.050 | 305.737 | 242.212 | 0.610 | 1.394 | 0.942 | 0.889 |
| 2 | 197.844 | 217.851 | 58923 | 76706 | 1064 | 140.296 | 70.106 | 192.361 | 28.585 | 2.744 | 280.592 | 140.212 | 384.723 | 318.244 | 274.128 | 0.508 | -0.099 | 0.768 | 0.654 |
| 3 | 536.210 | 240.238 | 35117 | 62792 | 1310 | 109.900 | 38.137 | 188.511 | 35.510 | 4.943 | 219.800 | 76.273 | 377.021 | 253.498 | 243.279 | 0.281 | 1.097 | 0.559 | 0.257 |
As três medidas principais (em unidades de pixel) são:
areaa área do objeto.area_cha área do casco convexo.perímetroo perímetro do objeto.
Usando essas medidas, a circularidade e a solidez são calculadas conforme mostrado em (Gupta et al, 2020).
$$ circularidade = 4 \pi(area / perimeter ^ 2) $$
$$ solidez = area / area \_ch$$
A circularidade é influenciada por serrilhas e saliências. A solidez é sensível a folhas com lóbulos profundos ou com pecíolo distinto e pode ser usada para distinguir folhas sem tais estruturas. Ao contrário da circularidade, não é muito sensível a serrilhas e saliências menores, uma vez que o casco convexo permanece praticamente inalterado.
Contorno do objeto
Os usuários também podem obter o contorno do objeto e o casco convexo da seguinte forma:
cont <-
object_contour(batata,
watershed = FALSE,
show_image = FALSE)
plot(batata)
plot_contour(cont, col = "red", lwd = 3)

Casco convexo
A função object_contour() retorna uma lista com os pontos de coordenadas para cada contorno do objeto que pode ser usado posteriormente para obter o casco convexo com conv_hull().
conv <- conv_hull(cont)
plot(batata)
plot_contour(conv, col = "black", lwd = 3)
plot_measures(pot_meas, measure = "solidity")

Área do casco convexo
Então, a área do casco convexo pode ser obtida com poly_area().
area <- poly_area(conv)
area
## $`1`
## [1] 54536
##
## $`2`
## [1] 76706
##
## $`3`
## [1] 62792.5
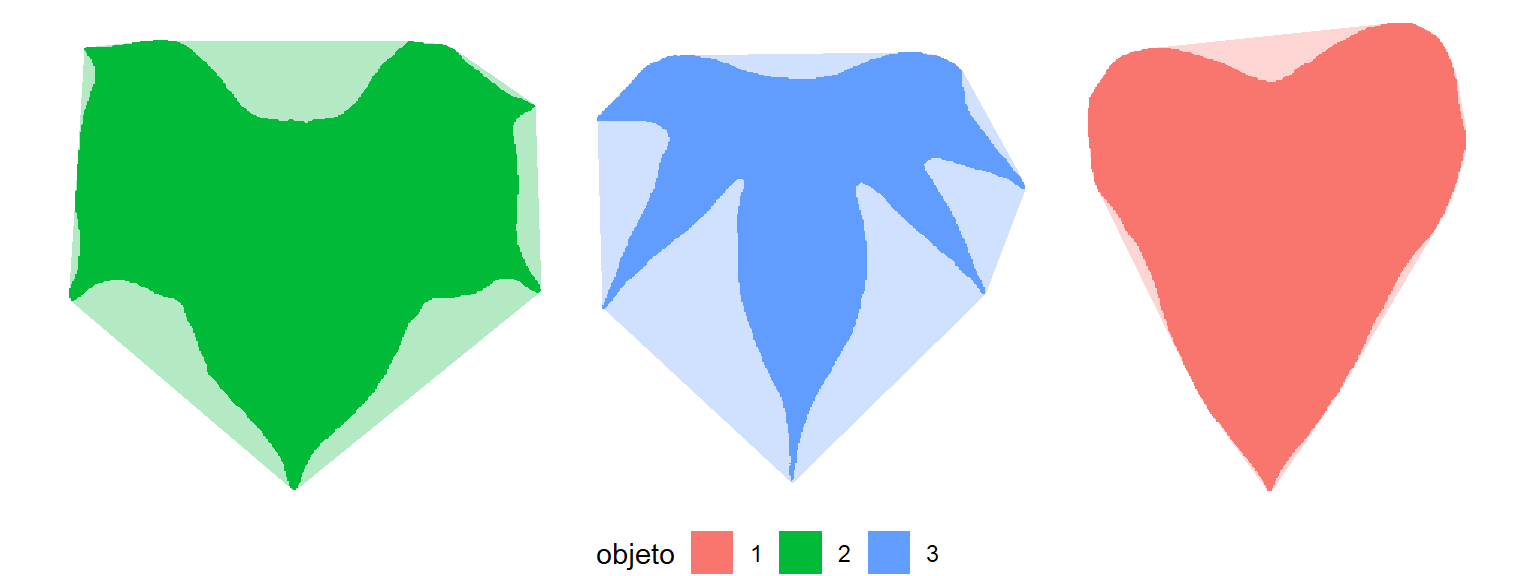
Folhas como ggplot2
# criar um quadro de dados para contorno e casco convexo
library(tidyverse)
df_cont <- bind_rows(cont, .id = "objeto")
df_conv <- bind_rows(conv, .id = "objeto")
ggplot(df_cont, aes(X1, X2, group = objeto)) +
geom_polygon(aes(fill = objeto)) +
geom_polygon(data = df_conv,
aes(x, y, fill = objeto),
alpha = 0.3) +
theme_void() +
theme(legend.position = "bottom")
Comprimento e largura
No exemplo a seguir, mostro como calcular o contorno do objeto, o centro de massa e os raios máximo e mínimo (em unidades de pixel). Nesse caso, o diâmetro mínimo e máximo (calculado com analyze_objects()) pode ser usado como uma medida para aproximar a largura e o comprimento do grão do feijão, respectivamente.
bean <- image_import("bean.jpg", plot = TRUE)

bean_meas <-
analyze_objects(bean,
index = "G",
fill_hull = TRUE,
watershed = FALSE,
show_contour = FALSE,
col_background = "black",
marker = "id")
bean_meas_cor <- get_measures(bean_meas, dpi = 300)
bean_meas_cor[, c("id", "diam_min", "diam_max")]
## id diam_min diam_max
## 1 1 0.998 2.272
## 6 6 0.916 2.324
## 11 11 1.002 2.105
## 12 12 0.940 2.151
## 29 29 0.875 1.867
## 34 34 0.940 2.248
# contorno
cont <-
object_contour(bean,
index = "G",
watershed = FALSE,
show_image = FALSE)
plot_contour(cont, col = "white", lwd = 2)
# centro de massa
cm <- poly_mass(cont)
plot_mass(cm,
col = "white",
arrow = TRUE)
# plota a largura e comprimento
plot_measures(bean_meas_cor, measure = "diam_min", vjust = 120)
plot_measures(bean_meas_cor, measure = "diam_max", hjust = 180)

Comprimento de raiz
No exemplo a seguir, mostro como medir o comprimento da raiz de mudas de soja. As imagens foram coletadas de Silva et al. 2019^[Silva LJ da, Medeiros AD de, Oliveira AMS (2019) SeedCalc, a new automated R software tool for germination and seedling length data processing. J Seed Sci 41:250–257. https://doi.org/10.1590/2317-1545V42N2217267︎].
roots <- image_import("root.jpg", plot = TRUE)
r1_meas <-
analyze_objects(roots,
index = "B",
marker = "id",
invert = TRUE)

Observe que a segmentação “watershed” segmentou os objetos conectados em vários objetos pequenos. Podemos melhorar esta segmentação usando o argumento tolerance (algumas tentativas serão necessárias para encontrar um valor adequado).
r1_meas <-
analyze_objects(roots,
index = "B",
marker = "id",
invert = TRUE,
tolerance = 3.5)
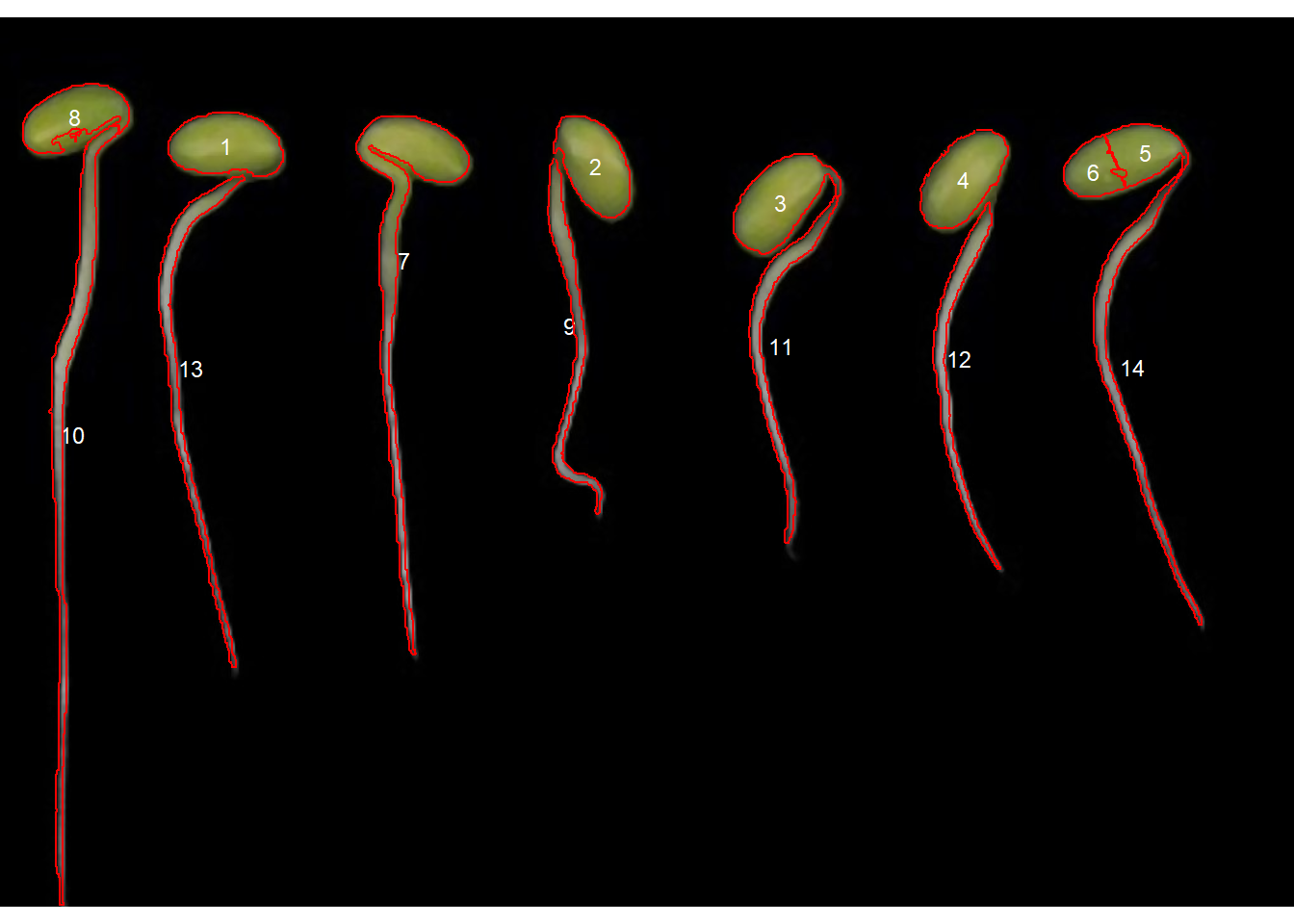
Muito melhor, mas ainda não é o que procuramos. Observe que as raízes e os cotilédones foram selecionados. Podemos, no entanto, usar uma restrição na seleção de objetos que, neste caso, pode ser a excentricidade do objeto. Usando os argumentos lower_eccent os objetos podem ser selecionados em relação à sua excentricidade.
r1_meas <-
analyze_objects(roots,
index = "B",
marker = "id",
invert = TRUE,
tolerance = 3,
lower_eccent = 0.95)
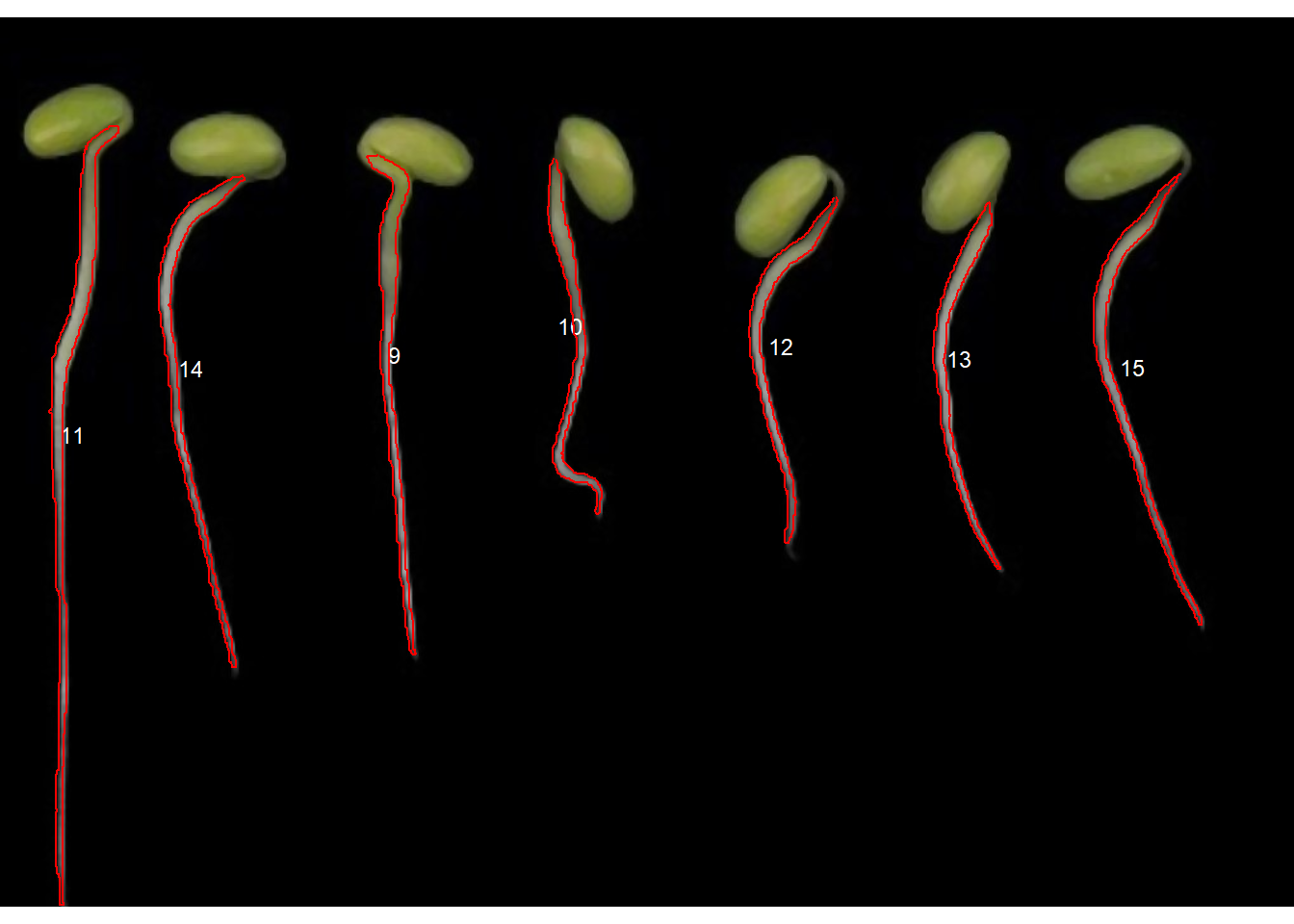
r1_meas_cor <- get_measures(r1_meas, dpi = 150)
# calcula a metade do perímetro
r1_meas_cor$metade_perimetro <- r1_meas_cor$perimeter / 2
# plota as medidas
plot(roots)
plot_measures(r1_meas_cor,
measure = "metade_perimetro",
hjust = 30,
col = "red")
plot_measures(r1_meas_cor,
measure = "diam_max",
hjust = -30)

Valores RGB para cada objeto
Para obter a intensidade RGB de cada objeto de imagem, usamos o argumento object_rgb = TRUE na função analyze_objects() . Neste exemplo,
img <- image_import("green.jpg", plot = TRUE)

# identifica o índice que melhor segmenta a imagem
image_binary(img, index = "all")

O índice NB (padrão) foi escolhido para segmentar os grãos do fundo. A média dos valores da banda verde será calculada declarando object_index = "G".
soy_green <-
analyze_objects(img,
object_index = "G",
marker_col = "black",
col_background = "white",
show_contour = FALSE)
plot_measures(soy_green,
measure = "G",
col = "black")

Parece que grãos com valores médios de verde (G) inferiores a 0.5 podem ser consideradas sementes esverdeadas. Os usuários podem então trabalhar com esse recurso e adaptá-lo ao seu caso.
report <- summary_index(soy_green, index = "G", cut_point = 0.5)
ids <- report$ids
plot(img)
plot_measures(soy_green,
id = ids,
measure = "G",
col = "black")
cont <- object_contour(img, show_image = FALSE)
plot_contour(cont, id = ids, col = "red")

Grande número de objetos
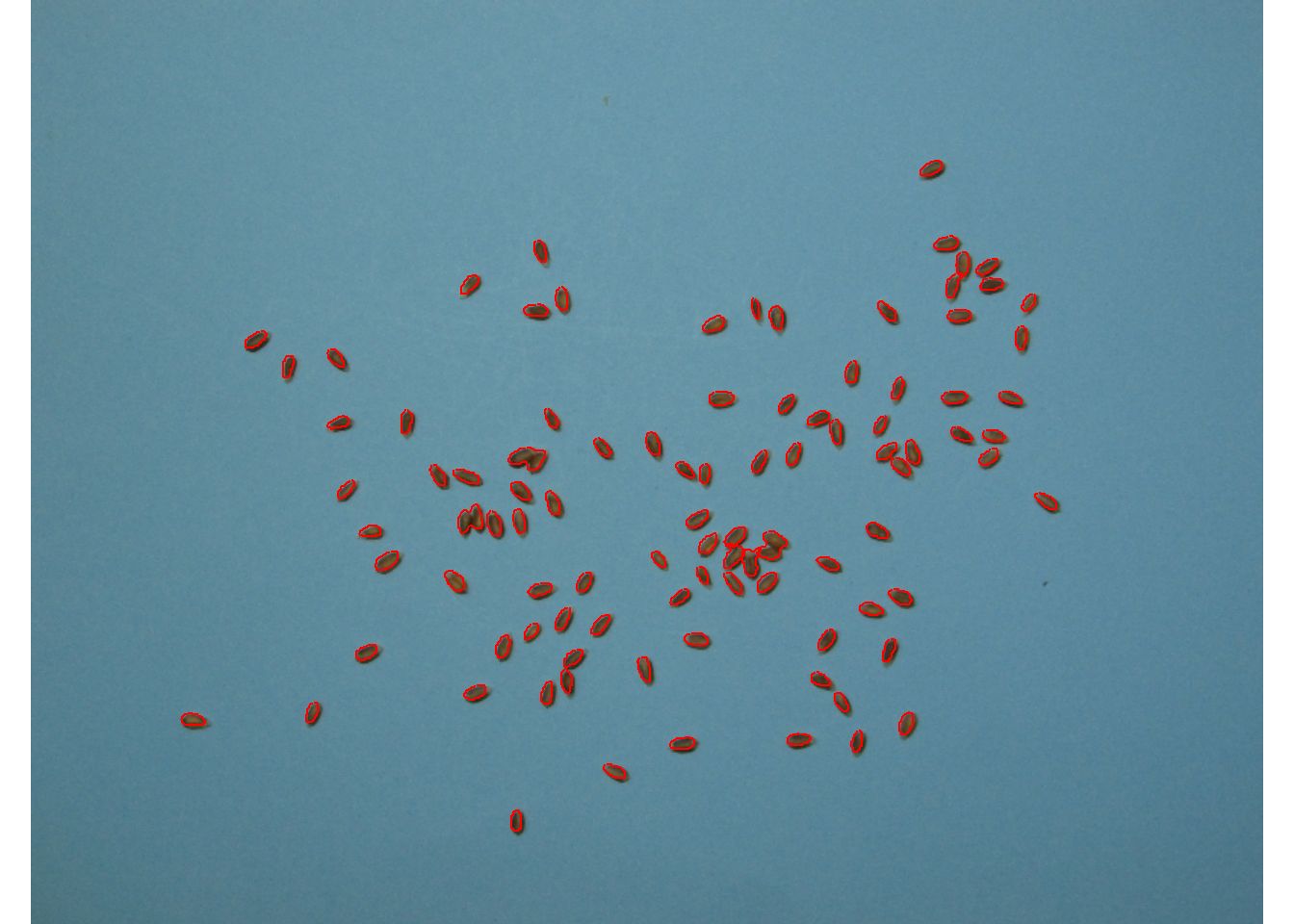
Quando existem muitos objetos, o argumento parallel = TRUE irá acelerar a extração dos valores RGB. No exemplo a seguir, uma imagem com 1343 grãos de Vicia cracca é analisada. Os índices "R" e "R/G" são computados. Os grãos com um valor médio de vermelho superior a 0,25 são destacados.
img2 <- image_import("vicia.jpg")
vicia <-
analyze_objects(img2,
index = "B",
object_index = pliman_indexes_eq(),
show_image = FALSE,
parallel = TRUE)
head(vicia$object_index) %>%
print_tbl()
| id | R | G | B | R/(R+G+B) | G/(R+G+B) | B/(R+G+B) | G/B | R/B | G/R | sqrt((R^2+G^2+B^2)/3) | sqrt((R2+G2+B*2)/3) | (R-G)/(R+G) | (2G-R-B)/(2G+R+B) | (2*R-G-B)/(G-B) | (G-R)/(G+R) | (G-B)/(G+B) | (R-B)/(R+B) | R+G+B | ((R+G+B)-3*B)/(R+G+B) | (G-R)/(G+R-B) | atan(2*(B-G-R)/30.5*(G-R)) | atan(2*(R-G-R)/30.5*(G-B)) | B/G | R+G+B/3 | 0.299R + 0.587G + 0.114*B | (25*(G-R)/(G+R-B)+1.25) | (max(R,G,B) - min(R,G,B)) / max(R,G,B) | (R-B)/R | 2*(R-G-B)/(G-B) | R2/(B*G3) |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 0.334 | 0.336 | 0.255 | 0.354 | 0.363 | 0.282 | 1.334 | 1.325 | 1.041 | 0.313 | 0.778 | -0.016 | 0.066 | -Inf | 0.016 | 0.132 | 0.115 | 0.925 | 0.153 | 5.000000e-02 | 0 | -0.002 | 0.782 | 0.755 | 0.326 | 2.509000e+00 | 0.912 | 0.168 | -Inf | 13.233 |
| 2 | 0.296 | 0.298 | 0.239 | 0.348 | 0.356 | 0.296 | 1.238 | 1.225 | Inf | 0.280 | 0.734 | -0.017 | 0.050 | -Inf | 0.017 | 0.097 | 0.081 | 0.833 | 0.111 | Inf | 0 | -0.001 | 0.836 | 0.673 | 0.290 | Inf | 1.000 | -Inf | -Inf | 19.327 |
| 3 | 0.281 | 0.289 | 0.229 | 0.348 | 0.362 | 0.290 | 1.263 | 1.223 | 1.045 | 0.268 | 0.725 | -0.020 | 0.062 | -Inf | 0.020 | 0.112 | 0.092 | 0.799 | 0.130 | 4.300000e-02 | 0 | -0.001 | 0.804 | 0.646 | 0.280 | 2.317000e+00 | 0.898 | 0.154 | -Inf | 15.999 |
| 4 | 0.290 | 0.300 | 0.240 | 0.345 | 0.361 | 0.294 | 1.253 | 1.207 | 1.058 | 0.279 | 0.736 | -0.026 | 0.061 | -Inf | 0.026 | 0.106 | 0.081 | 0.831 | 0.118 | 5.800000e-02 | 0 | -0.001 | 0.816 | 0.671 | 0.290 | 2.700000e+00 | 0.892 | 0.126 | -Inf | 16.655 |
| 5 | 0.270 | 0.301 | 0.256 | 0.319 | 0.364 | 0.317 | 1.181 | 1.060 | 1.230 | 0.278 | 0.738 | -0.074 | 0.068 | -Inf | 0.074 | 0.076 | 0.003 | 0.827 | 0.049 | 2.330000e-01 | 0 | -0.001 | 0.871 | 0.657 | 0.287 | 7.065000e+00 | 0.977 | -0.129 | -Inf | 10.694 |
| 6 | 0.154 | 0.202 | 0.246 | 0.255 | 0.335 | 0.410 | 0.825 | 0.640 | 1.531 | 0.206 | 0.630 | -0.147 | 0.005 | 3.018 | 0.147 | -0.097 | -0.235 | 0.603 | -0.229 | 6.872053e+12 | 0 | 0.001 | 1.220 | 0.439 | 0.193 | 1.718013e+14 | 0.992 | -0.974 | 14.723 | 14.218 |
cont2 <-
object_contour(img2,
index = "B",
show_image = FALSE)
ids2 <- which(vicia$object_index$R > 0.25)
plot(img2)
plot_contour(cont2, id = ids2, col = "red")

# cria gráfico de densidade dos valores RGB para as duas classes de grãos
rgbs <-
vicia$object_rgb %>%
mutate(type = ifelse(id %in% ids2, "Destacado", "Não destacado")) %>%
select(-id) %>%
pivot_longer(-type) %>%
subset(name == "G")
ggplot(rgbs, aes(x = value)) +
geom_density(fill = "green", alpha = 0.5) +
facet_wrap(~type)

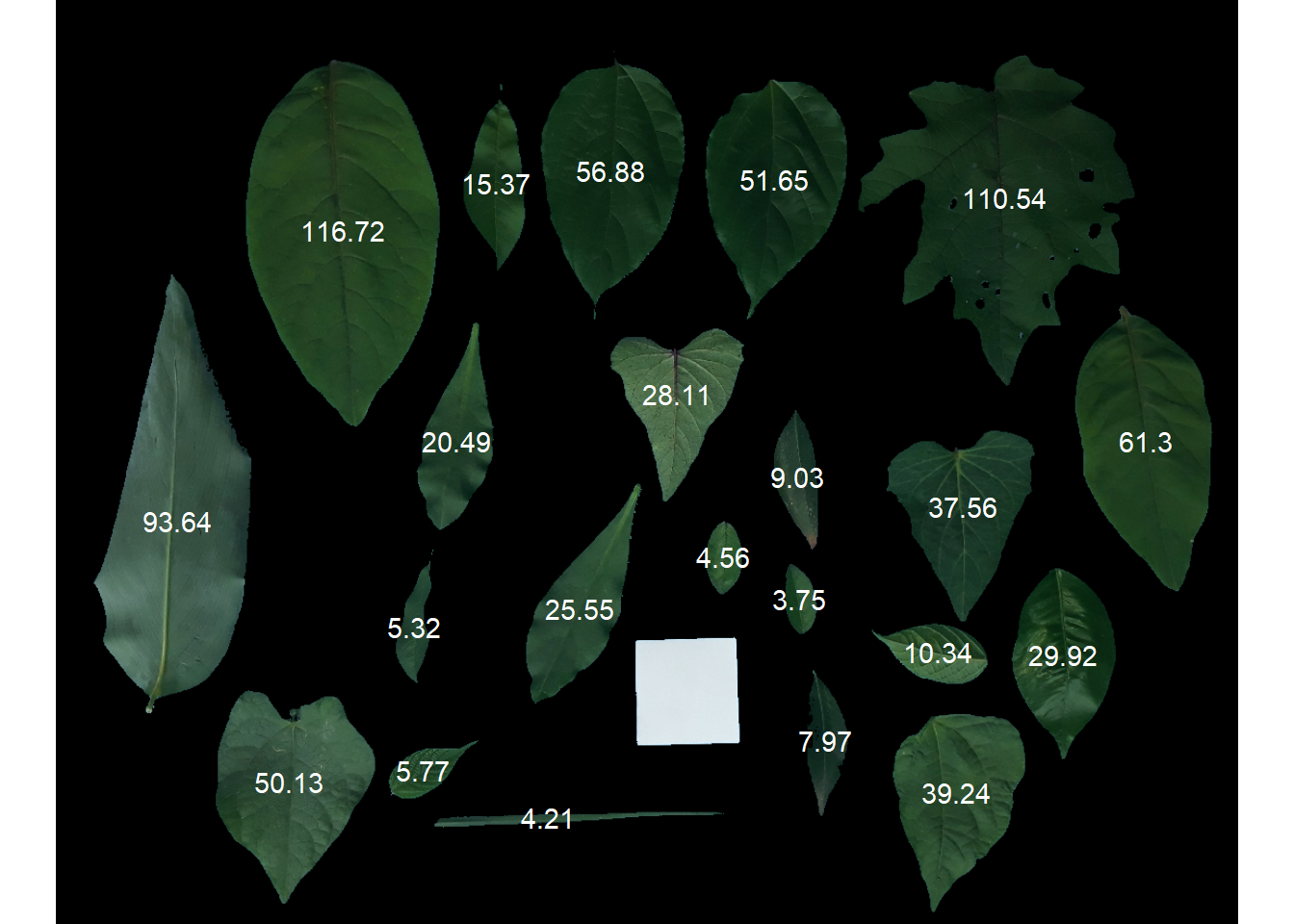
Área foliar
Uma imagem
folhas <- image_import(image = "leaves.jpg", plot = TRUE)

af <-
analyze_objects(folhas,
watershed = FALSE,
lower_eccent = 0.3,
show_contour = FALSE,
col_background = "black")
af_cor <- get_measures(af, dpi = 50.8)
plot_measures(af_cor, measure = "area")
Preenchendo ‘buracos’
Um aspecto importante a se considerar é quando há a presença de ‘buracos’ nas folhas. Isto pode ocorrer, por exemplo, pelo ataque de pragas. Neste caso, a área teria que ser considerada, pois ela estava lá!
holes <- image_import("holes.jpg", plot = TRUE)

af <-
analyze_objects(holes,
watershed = FALSE,
col_background = "white",
marker = "area",
marker_col = "red",
marker_size = 3,
show_image = FALSE,
save_image = TRUE,
dir_processed = tempdir(),
contour_size = 5)
af2 <-
analyze_objects(holes,
fill_hull = TRUE, # preenche 'buracos'
watershed = FALSE,
col_background = "white",
marker = "area",
marker_col = "red",
marker_size = 3,
show_image = FALSE,
save_image = TRUE,
prefix = "proc2_",
dir_processed = tempdir(),
contour_size = 5)
imgs <- image_import(pattern = "proc", path = tempdir())
image_combine(imgs)

Várias imagens da mesma amostra
Se os usuários precisarem analisar várias imagens da mesma amostra, as imagens da mesma amostra devem compartilhar o mesmo prefixo de nome de arquivo, que é definido como a parte do nome do arquivo que precede o primeiro hífen (-) ou underscore (_). Então, ao usar get_measures(), as medidas das imagens de folhas chamadas, por exemplo, F1-1.jpeg, F1_2.jpeg e F1-3.jpeg serão combinadas em uma única imagem (F1), mostrado no objeto merge. Isso é útil, por exemplo, para analisar folhas grandes que precisam ser divididas em várias imagens ou várias folhas pertencentes à mesma amostra que não podem ser digitalizadas em uma imagem única.
No exemplo a seguir, cinco imagens serão usadas como exemplos. Cada imagem possui folhas de diferentes espécies. As imagens foram divididas em imagens diferentes que compartilham o mesmo prefixo (por exemplo, L1_*, L2_* e assim por diante). Observe que para garantir que todas as imagens sejam processadas, todas as imagens devem compartilhar um padrão comum, neste caso (“L”). Os três pontos no canto inferior direito têm uma distância conhecida de 5 cm entre eles, que pode ser usada para extrair o dpi da imagem com dpi(). Apenas para fins didáticos, considerarei que todas as imagens têm resolução de 100 dpi.
# imagens inteiras
imgs <-
image_import(pattern = "leaf",
plot = TRUE,
ncol = 2)

# imagens da mesma amostra
sample_imgs <-
image_import(pattern = "L",
plot = TRUE,
ncol = 5)

Aqui, usarei o pattern =" L " para indicar que todas as imagens com este nome de padrão devem ser analisadas. O índice verde (" G ") é usado para segmentar as folhas e divisor de águas = FALSO é usado para omitir o algoritmo de segmentação de divisor de águas.
merged <-
analyze_objects(pattern = "L",
index = "B",
watershed = FALSE)
## Processing image L1_1 |==== | 8% 00:00:00

## Processing image L1_2 |======= | 17% 00:00:00

## Processing image L2_1 |========== | 25% 00:00:00

## Processing image L2_2 |============== | 33% 00:00:01

## Processing image L3_1 |================== | 42% 00:00:01

## Processing image L3_2 |===================== | 50% 00:00:01

## Processing image L3_3 |======================== | 58% 00:00:01

## Processing image L4_1 |============================ | 67% 00:00:02

## Processing image L4_2 |================================ | 75% 00:00:02

## Processing image L4_3 |=================================== | 83% 00:00:02

## Processing image L5_1 |====================================== | 92% 00:00:03

## Processing image L5_2 |==========================================| 100% 00:00:03
## --------------------------------------------
## Image Objects
## L1_1 1
## L1_2 1
## L2_1 2
## L2_2 3
## L3_1 1
## L3_2 1
## L3_3 1
## L4_1 2
## L4_2 2
## L4_3 3
## L5_1 3
## L5_2 3
## --------------------------------------------
## Done!

Usando a função get_measures() é possível converter as medidas de unidades de pixel em unidades métricas (cm$^ 2$).
merged_cor <- get_measures(merged, dpi = 100)
Observe que o merged_cor é uma lista com três objetos:
results: um data frame que contém as medidas de cada objeto individual (neste caso, folha) de cada imagem analisada.
merged_cor$results %>%
print_tbl()
| img | id | x | y | area | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| L1_1 | 1 | 427.723 | 516.719 | 102.170 | 292.253 | 115.189 | 6.217 | 0.561 | 11.913 | 114.118 | 21.226 | 12.435 | 1.122 | 23.826 | 19.863 | 15.144 | 0.647 | 0.158 | 0.350 | 0.097 |
| L1_2 | 1 | 372.556 | 526.031 | 92.840 | 207.023 | 107.188 | 5.341 | 1.042 | 10.168 | 86.064 | 9.754 | 10.682 | 2.085 | 20.336 | 15.123 | 13.215 | 0.486 | 0.600 | 0.448 | 0.102 |
| L2_1 | 1 | 251.404 | 282.798 | 46.319 | 46.789 | 24.003 | 3.936 | 2.527 | 5.920 | 35.873 | 2.342 | 7.872 | 5.054 | 11.840 | 10.479 | 5.708 | 0.839 | 1.502 | 0.990 | 1.010 |
| L2_1 | 2 | 146.366 | 701.900 | 35.305 | 36.136 | 21.488 | 3.441 | 2.311 | 5.209 | 32.274 | 2.254 | 6.881 | 4.621 | 10.417 | 9.308 | 4.873 | 0.852 | -1.533 | 0.977 | 0.961 |
| L2_2 | 1 | 326.113 | 391.141 | 19.026 | 19.221 | 16.739 | 2.613 | 1.659 | 4.129 | 29.010 | 2.488 | 5.225 | 3.319 | 8.259 | 7.301 | 3.375 | 0.887 | 1.560 | 0.990 | 0.853 |
| L2_2 | 2 | 113.850 | 755.321 | 38.153 | 38.670 | 20.904 | 3.534 | 2.518 | 5.097 | 26.159 | 2.024 | 7.069 | 5.036 | 10.195 | 8.955 | 5.462 | 0.792 | 1.556 | 0.987 | 1.097 |
| L2_2 | 3 | 376.213 | 784.804 | 16.320 | 16.439 | 14.580 | 2.365 | 1.532 | 3.616 | 23.766 | 2.361 | 4.730 | 3.063 | 7.231 | 6.509 | 3.238 | 0.867 | 1.395 | 0.993 | 0.965 |
| L3_1 | 1 | 253.962 | 480.290 | 64.082 | 117.849 | 79.324 | 4.199 | 0.408 | 9.476 | 80.727 | 23.207 | 8.397 | 0.817 | 18.952 | 15.225 | 8.681 | 0.822 | -1.550 | 0.544 | 0.128 |
| L3_2 | 1 | 200.077 | 436.484 | 30.205 | 57.570 | 54.813 | 2.830 | 0.065 | 6.576 | 56.098 | 100.588 | 5.661 | 0.131 | 13.152 | 9.857 | 7.103 | 0.693 | 1.564 | 0.525 | 0.126 |
| L3_3 | 1 | 204.984 | 363.476 | 52.026 | 87.382 | 80.137 | 3.494 | 0.077 | 7.821 | 65.208 | 101.216 | 6.989 | 0.155 | 15.641 | 12.404 | 8.414 | 0.735 | 1.538 | 0.595 | 0.102 |
| L4_1 | 1 | 270.258 | 326.544 | 54.355 | 54.773 | 24.638 | 4.177 | 3.390 | 5.717 | 21.940 | 1.686 | 8.354 | 6.780 | 11.434 | 9.918 | 7.035 | 0.705 | 1.484 | 0.992 | 1.125 |
| L4_1 | 3 | 252.952 | 845.157 | 61.679 | 63.975 | 28.499 | 4.540 | 3.654 | 6.488 | 27.727 | 1.776 | 9.080 | 7.307 | 12.975 | 10.549 | 7.549 | 0.699 | 1.428 | 0.964 | 0.954 |
| L4_2 | 1 | 291.942 | 235.565 | 61.503 | 62.870 | 28.067 | 4.544 | 3.148 | 6.592 | 32.407 | 2.094 | 9.087 | 6.297 | 13.183 | 10.970 | 7.278 | 0.748 | -1.332 | 0.978 | 0.981 |
| L4_2 | 2 | 260.787 | 799.754 | 73.268 | 75.388 | 30.658 | 4.938 | 3.787 | 7.013 | 30.654 | 1.852 | 9.876 | 7.574 | 14.026 | 11.609 | 8.152 | 0.712 | 1.527 | 0.972 | 0.980 |
| L4_3 | 1 | 206.166 | 213.417 | 29.186 | 29.581 | 19.279 | 3.097 | 2.054 | 4.374 | 26.315 | 2.130 | 6.193 | 4.107 | 8.748 | 8.256 | 4.535 | 0.836 | -1.536 | 0.987 | 0.987 |
| L4_3 | 2 | 219.514 | 552.896 | 19.503 | 20.439 | 16.713 | 2.595 | 1.608 | 3.940 | 25.493 | 2.451 | 5.191 | 3.215 | 7.881 | 7.069 | 3.548 | 0.865 | 1.538 | 0.954 | 0.877 |
| L4_3 | 3 | 229.163 | 937.157 | 34.149 | 35.426 | 23.520 | 3.470 | 2.211 | 5.138 | 31.462 | 2.324 | 6.940 | 4.421 | 10.277 | 8.809 | 5.090 | 0.816 | 1.471 | 0.964 | 0.776 |
| L5_1 | 1 | 225.338 | 275.720 | 52.797 | 54.448 | 31.217 | 4.590 | 2.259 | 7.735 | 63.899 | 3.425 | 9.179 | 4.517 | 15.470 | 13.610 | 5.029 | 0.929 | -1.479 | 0.970 | 0.681 |
| L5_1 | 2 | 335.525 | 884.869 | 31.675 | 32.608 | 24.460 | 3.619 | 1.727 | 6.199 | 52.019 | 3.590 | 7.237 | 3.454 | 12.398 | 10.718 | 3.823 | 0.934 | 1.382 | 0.971 | 0.665 |
| L5_1 | 3 | 120.360 | 887.853 | 30.192 | 32.323 | 27.102 | 3.827 | 1.649 | 6.774 | 59.974 | 4.109 | 7.654 | 3.297 | 13.549 | 11.055 | 3.579 | 0.946 | -1.564 | 0.934 | 0.517 |
| L5_2 | 1 | 339.022 | 300.133 | 45.622 | 46.825 | 30.023 | 4.331 | 2.188 | 7.465 | 63.358 | 3.412 | 8.662 | 4.376 | 14.930 | 13.160 | 4.466 | 0.941 | -1.547 | 0.974 | 0.636 |
| L5_2 | 2 | 205.390 | 802.624 | 42.866 | 43.898 | 29.464 | 4.267 | 2.060 | 7.335 | 62.330 | 3.561 | 8.534 | 4.120 | 14.670 | 12.766 | 4.355 | 0.940 | 1.508 | 0.976 | 0.621 |
| L5_2 | 3 | 535.301 | 819.600 | 44.996 | 46.294 | 29.337 | 4.300 | 1.992 | 7.465 | 62.372 | 3.748 | 8.600 | 3.984 | 14.930 | 13.069 | 4.450 | 0.940 | -1.450 | 0.972 | 0.657 |
summary: um data frame que contém o resumo dos resultados, contendo o número de objetos em cada imagem (n) a soma, média e desvio padrão da área de cada imagem, bem como o valor médio para todas as outras medidas (perímetro, raio, etc.)
merged_cor$summary %>%
print_tbl()
| img | n | area_sum | area_mean | area_sd | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| L1_1 | 1 | 102.170 | 102.170 | 0.000 | 292.253 | 115.189 | 6.217 | 0.561 | 11.913 | 114.118 | 21.226 | 12.435 | 1.122 | 23.826 | 19.863 | 15.144 | 0.647 | 0.158 | 0.350 | 0.097 |
| L1_2 | 1 | 92.840 | 92.840 | 0.000 | 207.023 | 107.188 | 5.341 | 1.042 | 10.168 | 86.064 | 9.754 | 10.682 | 2.085 | 20.336 | 15.123 | 13.215 | 0.486 | 0.600 | 0.448 | 0.102 |
| L2_1 | 2 | 81.624 | 40.812 | 7.788 | 41.462 | 22.746 | 3.688 | 2.419 | 5.564 | 34.073 | 2.298 | 7.377 | 4.838 | 11.128 | 9.893 | 5.290 | 0.845 | -0.016 | 0.983 | 0.986 |
| L2_2 | 3 | 73.500 | 24.500 | 11.901 | 24.777 | 17.407 | 2.837 | 1.903 | 4.281 | 26.312 | 2.291 | 5.675 | 3.806 | 8.562 | 7.588 | 4.025 | 0.849 | 1.504 | 0.990 | 0.972 |
| L3_1 | 1 | 64.082 | 64.082 | 0.000 | 117.849 | 79.324 | 4.199 | 0.408 | 9.476 | 80.727 | 23.207 | 8.397 | 0.817 | 18.952 | 15.225 | 8.681 | 0.822 | -1.550 | 0.544 | 0.128 |
| L3_2 | 1 | 30.205 | 30.205 | 0.000 | 57.570 | 54.813 | 2.830 | 0.065 | 6.576 | 56.098 | 100.588 | 5.661 | 0.131 | 13.152 | 9.857 | 7.103 | 0.693 | 1.564 | 0.525 | 0.126 |
| L3_3 | 1 | 52.026 | 52.026 | 0.000 | 87.382 | 80.137 | 3.494 | 0.077 | 7.821 | 65.208 | 101.216 | 6.989 | 0.155 | 15.641 | 12.404 | 8.414 | 0.735 | 1.538 | 0.595 | 0.102 |
| L4_1 | 2 | 116.034 | 58.017 | 5.178 | 59.374 | 26.568 | 4.358 | 3.522 | 6.102 | 24.833 | 1.731 | 8.717 | 7.044 | 12.205 | 10.233 | 7.292 | 0.702 | 1.456 | 0.978 | 1.040 |
| L4_2 | 2 | 134.771 | 67.385 | 8.319 | 69.129 | 29.362 | 4.741 | 3.468 | 6.802 | 31.530 | 1.973 | 9.482 | 6.936 | 13.605 | 11.289 | 7.715 | 0.730 | 0.097 | 0.975 | 0.980 |
| L4_3 | 3 | 82.838 | 27.613 | 7.449 | 28.482 | 19.837 | 3.054 | 1.957 | 4.484 | 27.757 | 2.302 | 6.108 | 3.915 | 8.968 | 8.045 | 4.391 | 0.839 | 0.491 | 0.968 | 0.880 |
| L5_1 | 3 | 114.664 | 38.221 | 12.645 | 39.793 | 27.593 | 4.012 | 1.878 | 6.903 | 58.631 | 3.708 | 8.023 | 3.756 | 13.806 | 11.795 | 4.144 | 0.937 | -0.554 | 0.958 | 0.621 |
| L5_2 | 3 | 133.484 | 44.495 | 1.445 | 45.672 | 29.608 | 4.299 | 2.080 | 7.422 | 62.687 | 3.573 | 8.599 | 4.160 | 14.843 | 12.998 | 4.424 | 0.940 | -0.496 | 0.974 | 0.638 |
merge: um data frame que contém os resultados mesclados pelo prefixo da imagem. Observe que, neste caso, os resultados são apresentados por L1, L2, L3, L4 e L5.
merged_cor$merge %>%
print_tbl()
| img | n | area_sum | area_mean | area_sd | area_ch | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | diam_mean | diam_min | diam_max | major_axis | minor_axis | eccentricity | theta | solidity | circularity |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| L1 | 2 | 195.010 | 97.505 | 0.000 | 249.638 | 111.189 | 5.779 | 0.802 | 11.040 | 100.091 | 15.490 | 11.558 | 1.604 | 22.081 | 17.493 | 14.179 | 0.567 | 0.379 | 0.399 | 0.099 |
| L2 | 5 | 155.124 | 32.656 | 9.845 | 33.120 | 20.077 | 3.263 | 2.161 | 4.922 | 30.192 | 2.295 | 6.526 | 4.322 | 9.845 | 8.741 | 4.658 | 0.847 | 0.744 | 0.987 | 0.979 |
| L3 | 3 | 146.313 | 48.771 | 0.000 | 87.600 | 71.425 | 3.508 | 0.184 | 7.958 | 67.344 | 75.004 | 7.016 | 0.367 | 15.915 | 12.495 | 8.066 | 0.750 | 0.517 | 0.555 | 0.119 |
| L4 | 7 | 333.643 | 51.005 | 6.982 | 52.328 | 25.256 | 4.051 | 2.982 | 5.796 | 28.040 | 2.002 | 8.102 | 5.965 | 11.592 | 9.856 | 6.466 | 0.757 | 0.681 | 0.974 | 0.967 |
| L5 | 6 | 248.149 | 41.358 | 7.045 | 42.733 | 28.600 | 4.156 | 1.979 | 7.162 | 60.659 | 3.641 | 8.311 | 3.958 | 14.325 | 12.396 | 4.284 | 0.938 | -0.525 | 0.966 | 0.629 |
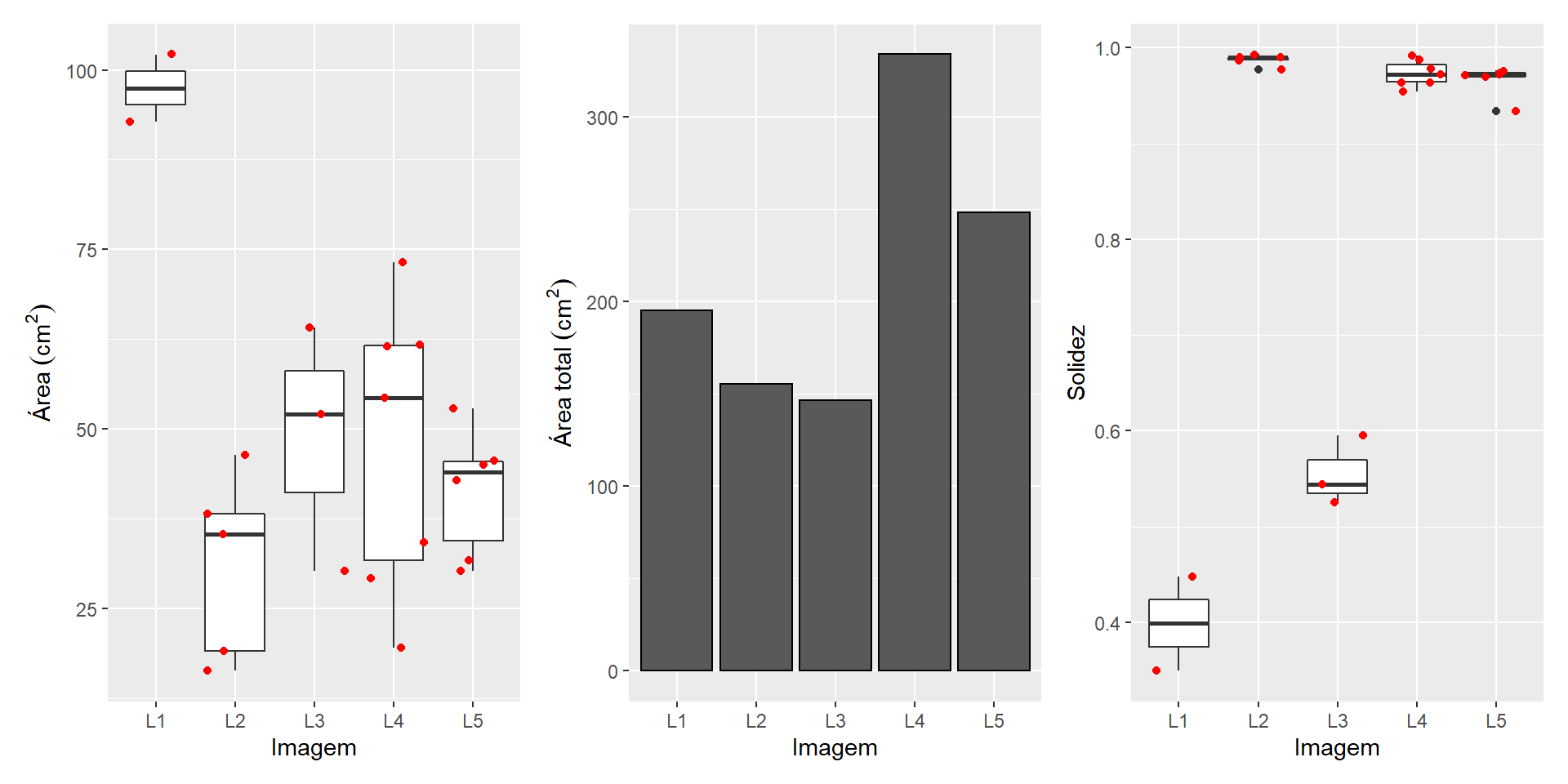
O area_sum de img L1 é a soma das duas folhas (uma em cada imagem)
sum(merged_cor$results$area[1:2])
## [1] 195.01
df_leaf <-
merged_cor$results %>%
separate(img, into = c("img", "code"))
# leaf area of the different species
p1 <-
ggplot(df_leaf, aes(x = img, y = area)) +
geom_boxplot() +
geom_jitter(color = "red") +
labs(x = "Imagem", y = expression(Área~(cm^2)))
p2 <-
ggplot(df_leaf, aes(x = img, y = area)) +
stat_summary(fun = sum,
geom = "bar",
col = "black") +
labs(x = "Imagem", y = expression(Área~total~(cm^2)))
# solidity of the different species
p3 <-
ggplot(df_leaf, aes(x = img, y = solidity)) +
geom_boxplot() +
geom_jitter(color = "red") +
labs(x = "Imagem", y = "Solidez")
p1 + p2 + p3 +
plot_layout(ncol = 3)