Fitopatometria
Diretório das imagens
# mudar de acordo com a pasta em seu computador
setwd("E:/Desktop/tiagoolivoto/static/tutorials/pliman_lca/imgs")
Severidade de doenças
Utilizando paletas
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
library(pliman)
## |==========================================================|
## | Tools for Plant Image Analysis (pliman 1.0.0) |
## | Author: Tiago Olivoto |
## | Type 'vignette('pliman_start')' for a short tutorial |
## | Visit 'http://bit.ly/pkg_pliman' for a complete tutorial |
## |==========================================================|
library(tidyverse)
## -- Attaching packages --------------------------------------- tidyverse 1.3.1 --
## v ggplot2 3.3.5 v purrr 0.3.4
## v tibble 3.1.6 v dplyr 1.0.7
## v tidyr 1.1.4 v stringr 1.4.0
## v readr 2.1.1 v forcats 0.5.1
## Warning: package 'tidyr' was built under R version 4.1.1
## Warning: package 'readr' was built under R version 4.1.2
## -- Conflicts ------------------------------------------ tidyverse_conflicts() --
## x forcats::%>%() masks stringr::%>%(), dplyr::%>%(), purrr::%>%(), tidyr::%>%(), tibble::%>%(), pliman::%>%()
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
img <- image_import("img_1.jpeg")
h <- image_import("h_img1.png")
d <- image_import("d_img1.png")
b <- image_import("b_img1.png")
image_combine(img, h, d, b, ncol = 4)

Gerando paletas
h2 <- pick_palette(img)
d2 <- pick_palette(img)
b2 <- pick_palette(img)
image_combine(h2, d2, b2, ncol = 3)
Padrão da função
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b)

Mostrando preenchimento das lesões
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_contour = FALSE)

Mostrando uma máscara
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_contour = FALSE,
show_original = FALSE)

Segmentando e analizando lesões
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
watershed = TRUE,
show_contour = FALSE, # não mostra os contornos
show_features = TRUE, # computa características das lesões
show_segmentation = TRUE) # mostra as segmentações

sev$severity
## healthy symptomatic
## 1 76.52184 23.47816
sev$statistics %>%
print_tbl()
| stat | value |
|---|---|
| n | 427.000 |
| min_area | 35.000 |
| mean_area | 418.028 |
| max_area | 6648.000 |
| sd_area | 561.783 |
| sum_area | 178498.000 |
sev$shape[1:10, ] %>%
print_tbl()
| id | x | y | area | perimeter | radius_mean | radius_min | radius_max | radius_sd | radius_ratio | major_axis | eccentricity | theta |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 1060.112 | 274.637 | 6648 | 394 | 46.660 | 19.898 | 72.080 | 11.628 | 3.623 | 118.273 | 0.686 | -0.036 |
| 2 | 1058.562 | 361.674 | 5326 | 369 | 40.613 | 24.949 | 53.431 | 6.464 | 2.142 | 90.033 | 0.433 | -1.508 |
| 3 | 463.503 | 536.277 | 6253 | 430 | 45.405 | 27.803 | 62.039 | 8.917 | 2.231 | 115.102 | 0.764 | -0.076 |
| 4 | 774.009 | 402.347 | 1745 | 166 | 23.292 | 18.509 | 30.490 | 2.932 | 1.647 | 54.225 | 0.625 | 1.066 |
| 5 | 800.022 | 317.335 | 2854 | 266 | 30.496 | 14.594 | 43.726 | 7.225 | 2.996 | 81.776 | 0.794 | 0.744 |
| 6 | 1123.083 | 351.937 | 2001 | 214 | 25.462 | 15.513 | 35.551 | 4.584 | 2.292 | 61.195 | 0.671 | -0.561 |
| 7 | 331.351 | 534.904 | 1202 | 138 | 19.263 | 12.376 | 24.908 | 2.732 | 2.013 | 45.366 | 0.629 | -1.562 |
| 8 | 1108.355 | 494.521 | 1078 | 116 | 18.259 | 14.763 | 24.157 | 2.119 | 1.636 | 38.820 | 0.303 | -1.133 |
| 9 | 1090.104 | 468.889 | 1185 | 136 | 19.392 | 9.087 | 25.175 | 4.068 | 2.771 | 48.675 | 0.715 | -1.013 |
| 10 | 788.304 | 363.604 | 1192 | 159 | 19.842 | 11.637 | 27.588 | 3.852 | 2.371 | 49.020 | 0.725 | -1.118 |
Utilizando índices RGB
Para identificar o melhor índice para segmentar a imagem do fundo e após as lesões da folha sadia, pode-se utilizar a função image_segment_iter. (OBS. somente funcionará em uma seção iterativa).
img %>%
image_resize(50) %>%
image_segment_iter(nseg = 2)
Após escolhidos os índices, podemos utilizar os argumentos index_lb e index_dh para segmentação da folha e fundo | lesão e sadio, respectivamente.
# após escolhidos os índices, utiliza
sev_index <-
measure_disease(img,
index_lb = "G",
index_dh = "NGRDI",
threshold = c("Otsu", -0.05),
show_image = TRUE)

Processamento em lote
Para analisar diversas imagens de um diretório, utiliza-se o argumento pattern, para declarar um padrão de nomes de arquivos. Serão utilizadas 20 folhas de soja disponíveis no repositório https://osf.io/4hbr6, um banco de dados de imagens de anotação de severidade de doenças de plantas. Obrigado a
Emerson M. Del Ponte e seus colaboradores por manter este projeto disponível publicamente.
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "healthy",
img_symptoms = "disease",
img_background = "back",
save_image = TRUE,
show_original = FALSE,
dir_processed = "processed",
show_image = FALSE)
)
## Processing image soy_1 |== | 5% 00:00:00
## Processing image soy_10 |==== | 10% 00:00:02
## Processing image soy_11 |====== | 15% 00:00:02
## Processing image soy_12 |======== | 20% 00:00:03
## Processing image soy_13 |========== | 25% 00:00:04
## Processing image soy_14 |============ | 30% 00:00:06
## Processing image soy_15 |============== | 35% 00:00:07
## Processing image soy_16 |================ | 40% 00:00:07
## Processing image soy_17 |================== | 45% 00:00:08
## Processing image soy_18 |==================== | 50% 00:00:10
## Processing image soy_19 |====================== | 55% 00:00:12
## Processing image soy_2 |========================= | 60% 00:00:14
## Processing image soy_20 |========================== | 65% 00:00:15
## Processing image soy_3 |============================= | 70% 00:00:17
## Processing image soy_4 |=============================== | 75% 00:00:17
## Processing image soy_5 |================================= | 80% 00:00:19
## Processing image soy_6 |=================================== | 85% 00:00:21
## Processing image soy_7 |===================================== | 90% 00:00:22
## Processing image soy_8 |======================================= | 95% 00:00:23
## Processing image soy_9 |=========================================| 100% 00:00:24
## usuário sistema decorrido
## 23.29 2.31 25.74
sev_lote$severity %>%
print_tbl()
| img | healthy | symptomatic |
|---|---|---|
| soy_1 | 93.316 | 6.684 |
| soy_10 | 85.335 | 14.665 |
| soy_11 | 16.849 | 83.151 |
| soy_12 | 66.002 | 33.998 |
| soy_13 | 79.255 | 20.745 |
| soy_14 | 62.275 | 37.725 |
| soy_15 | 58.377 | 41.623 |
| soy_16 | 50.488 | 49.512 |
| soy_17 | 82.083 | 17.917 |
| soy_18 | 53.107 | 46.893 |
| soy_19 | 87.621 | 12.379 |
| soy_2 | 63.575 | 36.425 |
| soy_20 | 51.526 | 48.474 |
| soy_3 | 62.815 | 37.185 |
| soy_4 | 44.305 | 55.695 |
| soy_5 | 84.058 | 15.942 |
| soy_6 | 78.668 | 21.332 |
| soy_7 | 76.380 | 23.620 |
| soy_8 | 70.316 | 29.684 |
| soy_9 | 54.912 | 45.088 |
Diagramas de área padrão
Os diagramas de área padrão (SAD) têm sido usados há muito tempo como uma ferramenta para auxiliar na estimativa da severidade de doenças de plantas, servindo como um modelo de referência padrão antes ou durante as avaliações.
Dado um objeto calculado com measure_disease(), um SAD com n imagens contendo os respectivos valores de severidade é obtido com sad().
As folhas com menor e maior severidade sempre estarão no SAD. Se n = 1, a folha com a menor severidade será retornada. As outras são amostradas sequencialmente para obter as n imagens após a severidade ter sido ordenada em ordem crescente. Por exemplo, se houver 30 folhas e n for definido como 3, as folhas amostradas serão a 1ª, 15ª e 30ª com os menores valores de severidade.
O SAD só pode ser calculado se um nome de padrão de imagem for usado no argumento pattern da função measure_disease(). Se as imagens forem salvas, as n imagens serão recuperadas do diretório dir_processed (diretório padrão por default). Caso contrário, a severidade será calculada novamente para gerar as imagens. Um SAD com 8 imagens do exemplo acima pode ser obtido facilmente com:
sad(sev_lote, n = 4, ncol = 4)
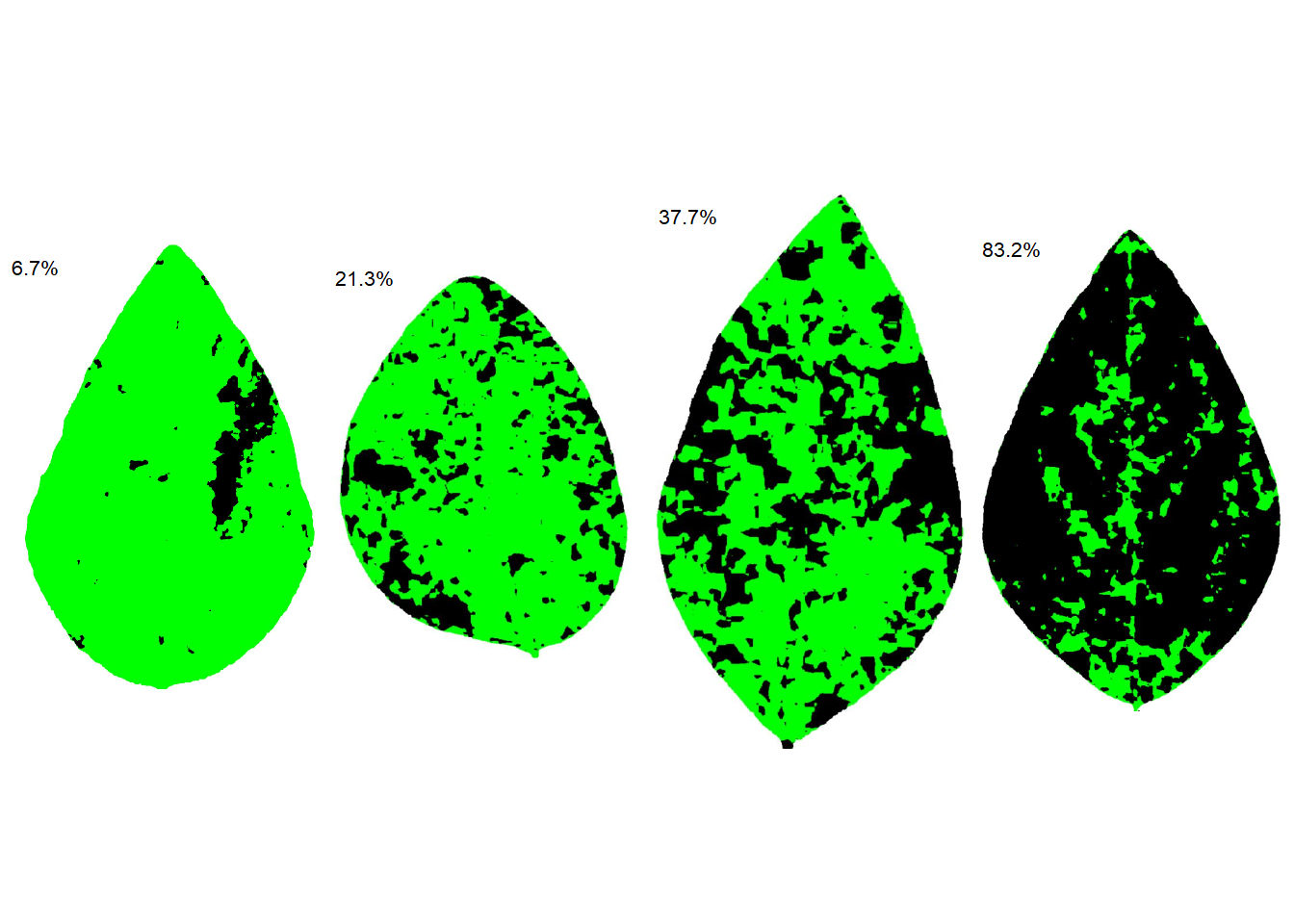
## img healthy symptomatic rank
## 1 soy_1 93.31565 6.684354 1
## 17 soy_6 78.66753 21.332469 7
## 6 soy_14 62.27506 37.724938 13
## 3 soy_11 16.84902 83.150984 20
Processamento paralelo
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "healthy",
img_symptoms = "disease",
img_background = "back",
show_image = FALSE,
parallel = TRUE)
)
## Image processing using multiple sessions (6). Please wait.
## usuário sistema decorrido
## 0.27 0.05 7.92
Imagens processadas
imgs <-
image_import(pattern = "proc_",
path = "processed",
plot = TRUE,
ncol = 5)

Um exemplo a mais
# criar paletas
img <- image_import( "multiplas_02.jpeg")
plot(img)

back <- pick_palette(img)
l <- pick_palette(img)
d <- pick_palette(img)
# usar as paletas na estimação de severidade
sev <-
measure_disease(pattern = "multip",
img_healthy = l,
img_symptoms = d,
img_background = back,
col_lesions = "red")
## Processing image multiplas_01 |======= | 20% 00:00:00

## Processing image multiplas_02 |============== | 40% 00:00:01

## Processing image multiplas_03 |==================== | 60% 00:00:02

## Processing image multiplas_04 |=========================== | 80% 00:00:02

## Processing image multiplas_05 |==================================| 100% 00:00:04

Número de ovos
O seguinte exemplo demonstra como contar o número de ovos em uma folha. A imagem foi obtida do pacote
ExpImage. Para identificar os melhores índices para segmentação da folha/fundo e ovos/folha, a função image_segment_iter() é usada.
ovos <- image_import("ovos.jpg", plot = TRUE)

# funciona apenas em uma seção iterativa
image_segment_iter(img, nseg = 2)
Após conhecer os índices, o número de ovos é computado com a função measure_disease(), indicando os índices para segmentação.
ovos_cont <-
measure_disease(ovos,
index_lb = "HUE2",
index_dh = "GRAY",
invert = c(FALSE, TRUE),
threshold = c("Otsu", 0.7),
show_features = TRUE,
show_segmentation = TRUE,
show_contour = FALSE)

ovos_cont$statistics
## stat value
## 1 n 170.000000
## 2 min_area 7.000000
## 3 mean_area 12.147059
## 4 max_area 21.000000
## 5 sd_area 2.085912
## 6 sum_area 2065.000000
ovos_cont <-
measure_disease(ovos,
index_lb = "HUE2",
index_dh = "GRAY",
invert = c(FALSE, TRUE),
threshold = c("Otsu", 0.7),
show_features = TRUE,
show_contour = FALSE,
col_lesions = "blue",
col_background = "black")

# apenas na versão de desenvolvimento
pick_count(ovos)
Mensuração iterativa
A função measure_disease_iter() fornece uma seção iterativa para measure_disease(), onde o usuário extrai amostras na imagem para criar as paletas de cores necessárias. measure_disease_iter() executa apenas em uma seção interativa. Nesta função, os usuários serão capazes extrair amostras de imagens para criar, iterativamente, as paletas de cores necessárias. Este processo chama pick_palette() internamente. Se has_background for TRUE (padrão), a paleta de cores do fundo é criada primeiro. A amostra de cores é realizada a cada clique com o botão esquerdo do mouse e continua até que o usuário pressione Esc. Em seguida, um novo processo de amostragem é realizado para amostrar a cor dos tecidos saudáveis e, em seguida, dos tecidos doentes. As paletas geradas são então passadas para measure_disease(). Todos os argumentos de tal função podem ser passados para measure_disease() usando ... (três pontos).
# somente roda em uma seção interativa
img <- image_pliman("sev_leaf.jpg", plot = TRUE)
measure_disease_iter(img)
Desafios
img <- image_import("maize_1.png", plot = TRUE)

image_segment(img, index = "all")

img <- image_import("maize_2.png", plot = TRUE)

image_segment(img, index = "all")

img2 <- image_crop(img,
width = 959:32,
height = 163:557,
plot = TRUE)
image_segment_iter(img2,
nseg = 2, # define o número de segmentações
index = c("NR", "GLI"), # índices para primeira e segunda
invert = c(T, F), # inverter a segmentação? (passa um vetor)
ncol = 3) # número de colunas no plot
## image pixels percent
## 1 original 1466240 100.00000
## 2 seg1 75147 5.12515
## 3 seg2 17325 23.05481
