Fitopatometria
Diretório das imagens
# mudar de acordo com a pasta em seu computador
setwd("E:/Desktop/tiagoolivoto/static/tutorials/pliman_ufsc_fito/leaves")
Severidade da doença
Utilizando paletas
library(pliman)
## |==========================================================|
## | Tools for Plant Image Analysis (pliman 1.1.0) |
## | Author: Tiago Olivoto |
## | Type 'citation('pliman')' to know how to cite pliman |
## | Type 'vignette('pliman_start')' for a short tutorial |
## | Visit 'http://bit.ly/pkg_pliman' for a complete tutorial |
## |==========================================================|
img <- image_import("exemp_1.jpeg", plot = TRUE)
h <- image_import("exem_h.png")
d <- image_import("exem_d.png")
b <- image_import("exem_b.png")
image_combine(img, h, d, b, ncol = 4)

Gerando paletas
h2 <- pick_palette(img)
d2 <- pick_palette(img)
b2 <- pick_palette(img)
image_combine(h2, d2, b2, ncol = 3)
Padrão da função
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b)

Mostrando preenchimento das lesões
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_contour = FALSE)

Mostrando uma máscara
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_contour = FALSE,
show_original = FALSE,
col_lesions = "brown") # padrão é "black"

Segmentando e analisando as lesões
Ao utilizar show_features = TRUE, a função analisa as lesões e retorna resultados como número de lesões, área, perímetro, etc. Com show_segmentation = TRUE, as lesões segmentadas são mostradas.
sev <-
measure_disease(img = img,
img_healthy = h,
img_symptoms = d,
img_background = b,
show_features = TRUE,
show_segmentation = TRUE)

# corrigir as medidas (dpi = 150)
sev_corrected <- get_measures(sev, dpi = 150)
Processamento em lote
Para analisar diversas imagens de um diretório, utiliza-se o argumento pattern, para declarar um padrão de nomes de arquivos. Serão utilizadas 15 folhas de soja disponíveis no repositório https://osf.io/4hbr6, um banco de dados de imagens de anotação de severidade de doenças de plantas. Obrigado a
Emerson M. Del Ponte e seus colaboradores por manter este projeto disponível publicamente.
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "soja_h",
img_symptoms = "soja_s",
img_background = "soja_b",
show_image = FALSE,
save_image = TRUE,
dir_processed = "processadas",
show_contour = FALSE,
col_lesions = "brown")
)
## Processing image soy_1 |= | 2% 00:00:00
## Processing image soy_10 |== | 4% 00:00:01
## Processing image soy_11 |== | 6% 00:00:03
## Processing image soy_12 |=== | 8% 00:00:04
## Processing image soy_13 |==== | 10% 00:00:05
## Processing image soy_14 |===== | 12% 00:00:06
## Processing image soy_15 |====== | 14% 00:00:07
## Processing image soy_16 |====== | 16% 00:00:08
## Processing image soy_17 |======= | 18% 00:00:09
## Processing image soy_18 |======== | 20% 00:00:10
## Processing image soy_19 |========= | 22% 00:00:12
## Processing image soy_2 |========== | 24% 00:00:13
## Processing image soy_20 |========== | 26% 00:00:13
## Processing image soy_21 |=========== | 28% 00:00:15
## Processing image soy_22 |============ | 30% 00:00:16
## Processing image soy_23 |============= | 32% 00:00:17
## Processing image soy_24 |============== | 34% 00:00:17
## Processing image soy_25 |============== | 36% 00:00:19
## Processing image soy_26 |=============== | 38% 00:00:20
## Processing image soy_27 |================ | 40% 00:00:21
## Processing image soy_28 |================= | 42% 00:00:23
## Processing image soy_29 |================== | 44% 00:00:24
## Processing image soy_3 |=================== | 46% 00:00:25
## Processing image soy_30 |=================== | 48% 00:00:26
## Processing image soy_31 |==================== | 50% 00:00:27
## Processing image soy_32 |===================== | 52% 00:00:27
## Processing image soy_33 |====================== | 54% 00:00:29
## Processing image soy_34 |====================== | 56% 00:00:30
## Processing image soy_35 |======================= | 58% 00:00:31
## Processing image soy_36 |======================== | 60% 00:00:32
## Processing image soy_37 |========================= | 62% 00:00:34
## Processing image soy_38 |========================== | 64% 00:00:35
## Processing image soy_39 |========================== | 66% 00:00:36
## Processing image soy_4 |============================ | 68% 00:00:37
## Processing image soy_40 |============================ | 70% 00:00:38
## Processing image soy_41 |============================= | 72% 00:00:38
## Processing image soy_42 |============================== | 74% 00:00:40
## Processing image soy_43 |============================== | 76% 00:00:41
## Processing image soy_44 |=============================== | 78% 00:00:42
## Processing image soy_45 |================================ | 80% 00:00:43
## Processing image soy_46 |================================= | 82% 00:00:44
## Processing image soy_47 |================================== | 84% 00:00:45
## Processing image soy_48 |================================== | 86% 00:00:46
## Processing image soy_49 |=================================== | 88% 00:00:47
## Processing image soy_5 |===================================== | 90% 00:00:48
## Processing image soy_50 |===================================== | 92% 00:00:49
## Processing image soy_6 |======================================= | 94% 00:00:50
## Processing image soy_7 |======================================= | 96% 00:00:50
## Processing image soy_8 |======================================== | 98% 00:00:51
## Processing image soy_9 |=========================================| 100% 00:00:52
## usuário sistema decorrido
## 50.09 3.28 53.45
sev_lote$severity
## img healthy symptomatic
## 1 soy_1 92.59873 7.4012695
## 2 soy_10 53.35002 46.6499823
## 3 soy_11 87.96519 12.0348127
## 4 soy_12 62.66145 37.3385524
## 5 soy_13 50.30860 49.6913960
## 6 soy_14 99.70519 0.2948133
## 7 soy_15 71.34672 28.6532822
## 8 soy_16 30.96793 69.0320692
## 9 soy_17 22.52065 77.4793520
## 10 soy_18 83.18994 16.8100553
## 11 soy_19 40.48431 59.5156887
## 12 soy_2 85.48984 14.5101631
## 13 soy_20 34.78690 65.2130957
## 14 soy_21 33.33936 66.6606360
## 15 soy_22 75.07775 24.9222506
## 16 soy_23 58.57313 41.4268726
## 17 soy_24 74.88339 25.1166105
## 18 soy_25 10.08844 89.9115622
## 19 soy_26 28.94710 71.0528969
## 20 soy_27 33.14847 66.8515320
## 21 soy_28 51.49431 48.5056892
## 22 soy_29 22.87381 77.1261863
## 23 soy_3 16.15317 83.8468283
## 24 soy_30 44.41465 55.5853493
## 25 soy_31 14.40058 85.5994179
## 26 soy_32 45.90498 54.0950186
## 27 soy_33 89.28918 10.7108200
## 28 soy_34 43.01467 56.9853341
## 29 soy_35 57.40314 42.5968600
## 30 soy_36 94.80323 5.1967658
## 31 soy_37 35.66992 64.3300826
## 32 soy_38 52.14612 47.8538833
## 33 soy_39 40.87600 59.1239978
## 34 soy_4 66.79356 33.2064430
## 35 soy_40 68.16094 31.8390630
## 36 soy_41 97.29642 2.7035831
## 37 soy_42 85.94240 14.0576023
## 38 soy_43 90.80627 9.1937327
## 39 soy_44 58.67789 41.3221098
## 40 soy_45 83.40446 16.5955434
## 41 soy_46 84.17359 15.8264079
## 42 soy_47 76.98019 23.0198057
## 43 soy_48 76.29143 23.7085736
## 44 soy_49 70.82702 29.1729836
## 45 soy_5 77.01014 22.9898597
## 46 soy_50 54.78424 45.2157630
## 47 soy_6 63.73643 36.2635748
## 48 soy_7 58.39910 41.6008962
## 49 soy_8 40.69830 59.3016995
## 50 soy_9 78.37495 21.6250451
Diagramas de área padrão
Os diagramas de área padrão (SAD) têm sido usados há muito tempo como uma ferramenta para auxiliar na estimativa da severidade de doenças de plantas, servindo como um modelo de referência padrão antes ou durante as avaliações.
Dado um objeto calculado com measure_disease(), um SAD com n imagens contendo os respectivos valores de severidade é obtido com sad().
As folhas com menor e maior severidade sempre estarão no SAD. Se n = 1, a folha com a menor severidade será retornada. As outras são amostradas sequencialmente para obter as n imagens após a severidade ter sido ordenada em ordem crescente. Por exemplo, se houver 30 folhas e n for definido como 3, as folhas amostradas serão a 1ª, 15ª e 30ª com os menores valores de severidade.
O SAD só pode ser calculado se um nome de padrão de imagem for usado no argumento pattern da função measure_disease(). Se as imagens forem salvas, as n imagens serão recuperadas do diretório dir_processed (diretório padrão por default). Caso contrário, a severidade será calculada novamente para gerar as imagens. Um SAD com 8 imagens do exemplo acima pode ser obtido facilmente com:
sad(sev_lote, n = 6, ncol = 3)

## img healthy symptomatic rank
## 6 soy_14 99.70519 0.2948133 1
## 41 soy_46 84.17359 15.8264079 10
## 44 soy_49 70.82702 29.1729836 20
## 2 soy_10 53.35002 46.6499823 30
## 31 soy_37 35.66992 64.3300826 40
## 18 soy_25 10.08844 89.9115622 50
Processamento paralelo
Para acelerar o tempo de processamento quando várias imagens estão disponíveis, pode-se utilizar o argumento paralell. Isto criará múltiplas seções R em segundo plano, sendo cada uma responsável pelo processamento de uma parte das imagens.
system.time(
sev_lote <-
measure_disease(pattern = "soy",
img_healthy = "soja_h",
img_symptoms = "soja_s",
img_background = "soja_b",
show_image = FALSE,
parallel = TRUE)
)
## Image processing using multiple sessions (6). Please wait.
## usuário sistema decorrido
## 0.20 0.06 19.12
No próximo exemplo, são analisadas oito folhas de tomateiro. As imagens são salvas em um arquivo temporário e posteriormente importadas.
dir <- tempdir()
sev_tomate <-
measure_disease(pattern = "tomate_",
img_healthy = "tom_h",
img_symptoms = "tom_s",
img_background = "tom_b",
col_lesions = "red",
show_contour = FALSE,
save_image = TRUE,
dir_processed = dir,
parallel = TRUE)
## Image processing using multiple sessions (6). Please wait.
a <-
image_import(pattern = "proc_",
path = dir,
plot = TRUE,
ncol = 4)

Várias imagens da mesma amostra
Se os usuários precisarem analisar várias imagens da mesma amostra, as imagens da mesma amostra devem compartilhar o mesmo prefixo de nome de arquivo, que é definido como a parte do nome do arquivo que precede o primeiro hífen (-) ou underscore (_).
No exemplo a seguir, 16 imagens serão usadas como exemplos. Aqui, elas representam quatro repetiçoes de quatro diferentes tratamentos (TRAT1_1, TRAT1_2, ..., TRAT4_4). Observe que para garantir que todas as imagens sejam processadas, todas as imagens devem compartilhar um padrão comum, neste caso (“TRAT”).
system.time(
sev_trats <-
measure_disease(pattern = "TRAT",
img_healthy = "feijao_h",
img_symptoms = "feijao_s",
img_background = "feijao_b",
show_features = TRUE,
show_image = FALSE,
parallel = TRUE)
)
## Image processing using multiple sessions (6). Please wait.
## usuário sistema decorrido
## 0.25 0.06 10.30
sev <-
sev_trats$severity |>
separate_col(img, into = c("TRAT", "REP"))
library(ggplot2)
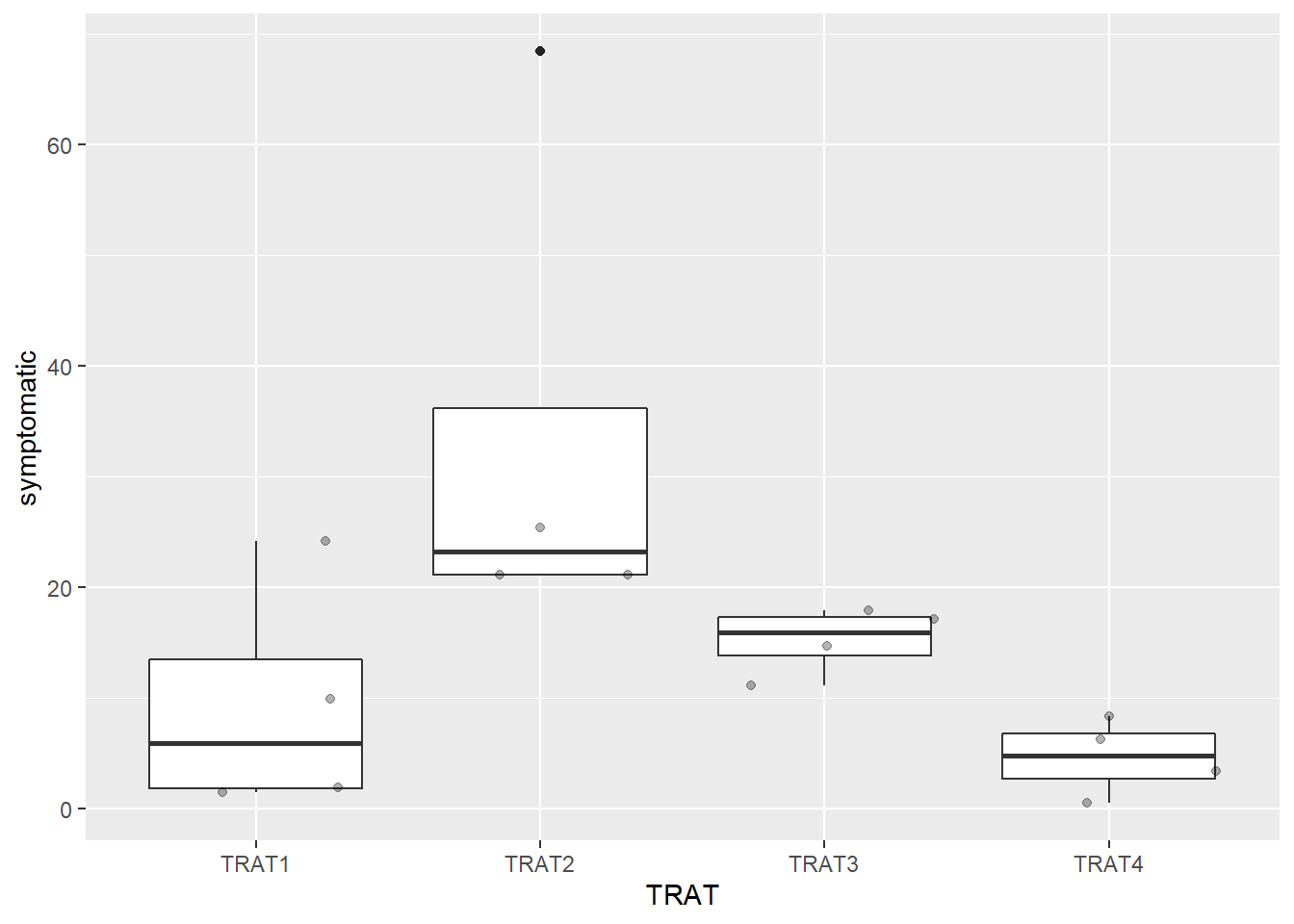
ggplot(sev, aes(TRAT, symptomatic))+
geom_boxplot() +
geom_jitter(alpha = 0.3)
Múltiplas folhas em uma foto
Quando múltiplas folhas estão presentes em uma imagem, a função measure_disease retorna a severidade média das folhas presentes na imagem. Para quantificar a severidade por folha, a função measure_disease_byl() pode ser utilizada.
Esta função calcula a porcentagem de área foliar sintomática usando paletas de cores ou índices RGB para cada folha (byl) de uma imagem. Isso permite, por exemplo, processar réplicas do mesmo tratamento e obter os resultados de cada replicação com uma única imagem. Para fazer isso, as amostras de folhas são primeiro divididas usando a função object_split() e, em seguida, a função measure_disease() é aplicado à lista de folhas.
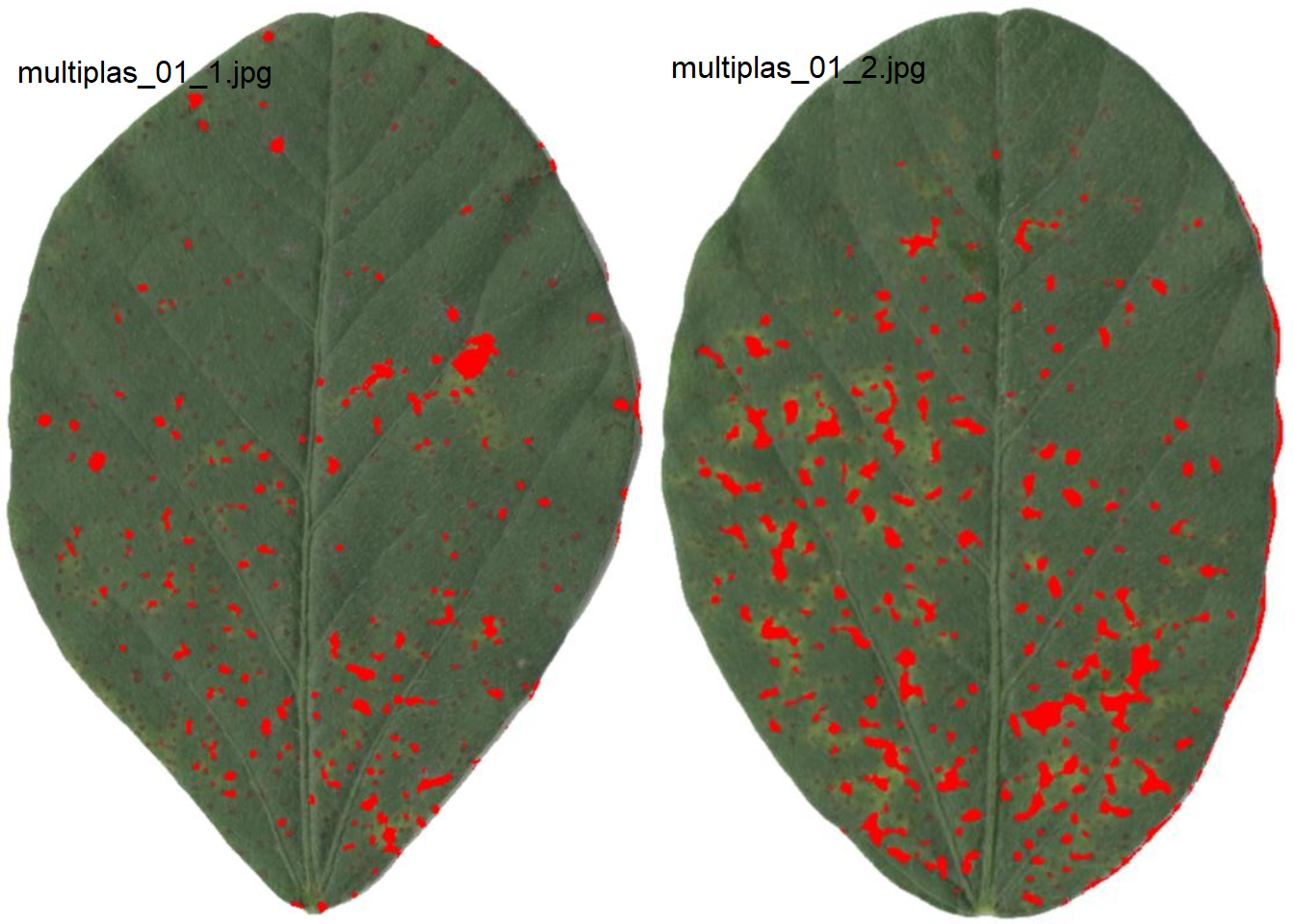
mult1 <- image_import("multiplas_01.jpeg", plot = TRUE)

image_index(mult1)

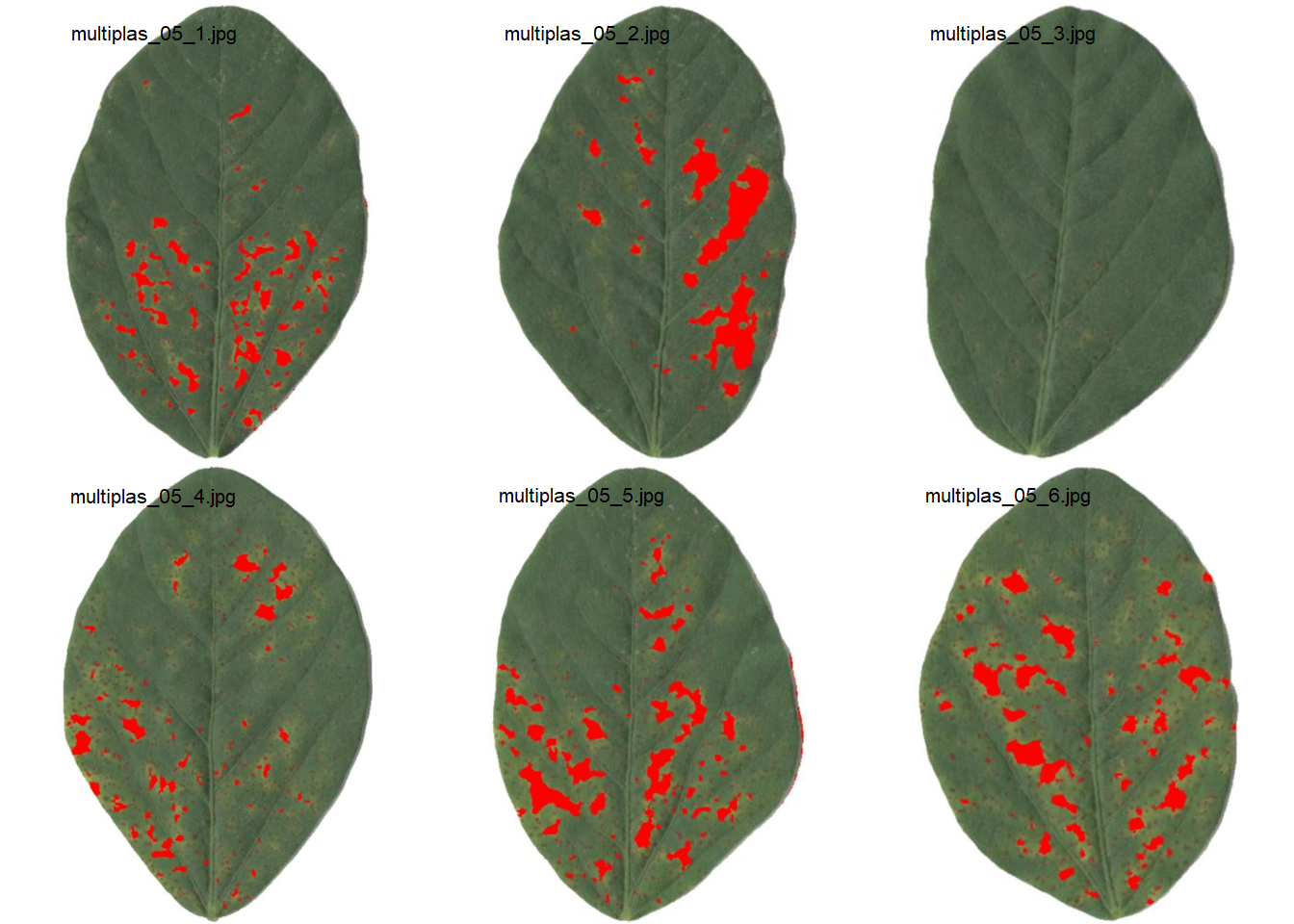
byl <-
measure_disease_byl(pattern = "multiplas_",
index = "B", # usado para segmentar a folha do fundo
img_healthy = "soja_h",
img_symptoms = "soja_s",
show_contour = FALSE,
show_features = TRUE,
col_lesions = "red")
## Processing image multiplas_01 |======= | 20% 00:00:00
## Processing image multiplas_02 |============== | 40% 00:00:02
## Processing image multiplas_03 |==================== | 60% 00:00:06

## Processing image multiplas_04 |=========================== | 80% 00:00:10

## Processing image multiplas_05 |==================================| 100% 00:00:13

results_byl <- get_measures(byl)
head(results_byl$results)
## img leaf id x y area area_ch perimeter radius_mean
## 1 multiplas_01 1 1 231.377 27.082 61 53 23 3.977
## 2 multiplas_01 1 2 374.197 30.974 76 68 32 4.798
## 3 multiplas_01 1 6 317.000 62.000 5 0 8 1.000
## 4 multiplas_01 1 9 168.271 82.136 118 108 37 5.712
## 5 multiplas_01 1 10 226.706 86.294 17 10 12 1.894
## 6 multiplas_01 1 11 175.250 104.028 36 27 19 3.039
## radius_min radius_max radius_sd radius_ratio diam_mean diam_min diam_max
## 1 2.943 4.654 0.420 1.582 7.954 5.885 9.308
## 2 1.971 7.425 1.437 3.768 9.596 3.941 14.850
## 3 0.000 2.000 0.707 Inf 2.000 0.000 4.000
## 4 3.561 7.541 0.953 2.118 11.424 7.122 15.083
## 5 1.294 2.647 0.394 2.046 3.788 2.587 5.294
## 6 2.003 4.520 0.806 2.257 6.077 4.006 9.040
## major_axis minor_axis eccentricity theta solidity circularity
## 1 9.154 8.653 0.326 -0.970 1.151 1.449
## 2 14.535 7.377 0.862 0.695 1.118 0.933
## 3 5.657 0.000 1.000 1.571 Inf 0.982
## 4 13.596 11.943 0.478 -1.318 1.093 1.083
## 5 5.552 3.917 0.709 -0.459 1.700 1.484
## 6 9.225 5.175 0.828 0.998 1.333 1.253
head(results_byl$summary)
## img n area_sum area_mean area_sd area area_ch perimeter
## 1 multiplas_01 450 202897.49 450.883 159.524 54.149 58.424 24.218
## 2 multiplas_02 320 133775.99 418.050 171.943 387.172 587.731 71.338
## 3 multiplas_03 408 148768.67 364.629 150.484 160.716 225.512 40.181
## 4 multiplas_04 184 83653.87 454.641 145.914 146.495 199.467 36.076
## 5 multiplas_05 340 116878.23 343.760 115.416 123.703 155.224 35.694
## radius_mean radius_min radius_max radius_sd radius_ratio diam_mean diam_min
## 1 3.418 1.371 5.341 1.138 Inf 6.836 2.742
## 2 8.116 2.554 13.166 2.746 8.579 16.232 5.108
## 3 5.019 1.878 8.037 1.651 Inf 10.037 3.756
## 4 4.529 1.571 7.274 1.542 Inf 9.058 3.142
## 5 4.652 1.705 7.336 1.526 7.322 9.304 3.411
## diam_max major_axis minor_axis eccentricity theta solidity circularity
## 1 10.682 11.217 5.387 0.777 0.095 Inf 1.280
## 2 26.332 25.276 13.245 0.787 -0.029 1.052 0.900
## 3 16.074 15.837 8.236 0.771 0.052 1.306 1.142
## 4 14.547 14.715 7.187 0.796 0.132 Inf 1.332
## 5 14.672 14.632 7.655 0.768 0.092 Inf 1.215
head(results_byl$merge)
## img n area_sum area_mean area_sd area area_ch perimeter
## 1 multiplas 1702 685974.2 406.392 148.656 174.447 245.272 41.501
## radius_mean radius_min radius_max radius_sd radius_ratio diam_mean diam_min
## 1 5.147 1.816 8.231 1.72 Inf 10.293 3.632
## diam_max major_axis minor_axis eccentricity theta solidity circularity
## 1 16.461 16.335 8.342 0.78 0.068 Inf 1.174