Estatísticas descritivas
O metan fornece uma estrutura simples e intuitiva, compatível com o pipe, para realizar estatísticas descritivas. Um
conjunto de funções pode ser usado para calcular rapidamente as estatísticas descritivas mais utilizadas.
library(metan)
library(rio)
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
# dados
df <- import("http://bit.ly/df_ok", setclass = "tbl")
inspect(df, verbose = FALSE) %>% print_tbl()
| Variable | Class | Missing | Levels | Valid_n | Min | Median | Max | Outlier |
|---|---|---|---|---|---|---|---|---|
| ENV | character | No | 0 | 114 | NA | NA | NA | NA |
| GEN | character | No | 0 | 114 | NA | NA | NA | NA |
| BLOCO | character | No | 0 | 114 | NA | NA | NA | NA |
| ALT_PLANT | numeric | Yes | - | 113 | 1.71 | 2.52 | 3.04 | 0 |
| ALT_ESP | numeric | No | - | 114 | 0.75 | 1.34 | 1.88 | 0 |
| COMPES | numeric | No | - | 114 | 12.24 | 15.10 | 17.94 | 0 |
| DIAMES | numeric | No | - | 114 | 43.48 | 49.84 | 54.86 | 0 |
| COMP_SAB | numeric | No | - | 114 | 23.49 | 28.25 | 34.66 | 0 |
| DIAM_SAB | numeric | No | - | 114 | 13.28 | 16.03 | 18.28 | 0 |
| MGE | numeric | No | - | 114 | 105.72 | 171.74 | 250.89 | 0 |
| NFIL | numeric | No | - | 114 | 12.40 | 16.00 | 21.20 | 1 |
| MMG | numeric | No | - | 114 | 226.60 | 336.05 | 451.68 | 0 |
| NGE | numeric | Yes | - | 112 | 354.00 | 504.40 | 696.60 | 6 |
Vamos começar com um exemplo muito simples (mas amplamente usado): calcular a média de uma variável numérica (digamos, MGE) do conjunto de dados df. Usando a função R base stats::mean(), a solução seria:
mean(df$MGE)
## [1] 170.7426
Considere que agora queremos calcular o valor médio da MGE para cada nível do fator GEN. Em outras palavras, calcular o valor médio da MGE para cada genótipo. A solução usando stats::aggregate() é então:
aggregate(MGE ~ GEN, data = df, FUN = mean) %>% print_tbl()
| GEN | MGE |
|---|---|
| H1 | 172.507 |
| H10 | 157.621 |
| H11 | 164.192 |
| H12 | 153.373 |
| H13 | 189.759 |
| H2 | 194.332 |
| H3 | 176.047 |
| H4 | 180.913 |
| H5 | 180.021 |
| H6 | 194.650 |
| H7 | 160.334 |
| H8 | 150.167 |
| H9 | 146.326 |
Estatísticas por níveis de um fator
Usando a função means_by() a quantidade de código necessária é drasticamente reduzida. Para calcular a média geral para todas as variáveis numéricas de df, simplesmente usamos:
ov_mean <- means_by(df)
## Warning: NA values removed to compute the function. Use 'na.rm = TRUE' to
## suppress this warning.
## To remove rows with NA use `remove_rows_na()'.
## To remove columns with NA use `remove_cols_na()'.
print_tbl(ov_mean)
| ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|
| 2.463 | 1.314 | 15.167 | 49.362 | 28.901 | 16.026 | 170.743 | 16.154 | 336.168 | 508.018 |
Para calcular os valores médios para cada nível do fator GEN, precisamos adicionar a variável de agrupamento GEN no argumento ...
means_gen <- means_by(df, GEN)
## Warning: NA values removed to compute the function. Use 'na.rm = TRUE' to
## suppress this warning.
## To remove rows with NA use `remove_rows_na()'.
## To remove columns with NA use `remove_cols_na()'.
print_tbl(means_gen)
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 2.563 | 1.445 | 14.903 | 51.232 | 31.205 | 15.650 | 172.507 | 16.467 | 378.852 | 454.767 |
| H10 | 2.220 | 1.206 | 15.204 | 47.926 | 27.644 | 16.092 | 157.621 | 15.333 | 312.234 | 504.933 |
| H11 | 2.331 | 1.233 | 15.333 | 47.763 | 27.522 | 16.076 | 164.192 | 15.067 | 327.879 | 488.850 |
| H12 | 2.407 | 1.243 | 14.166 | 48.162 | 27.624 | 14.727 | 153.373 | 16.178 | 309.880 | 498.067 |
| H13 | 2.531 | 1.332 | 15.293 | 51.468 | 30.139 | 16.073 | 189.759 | 18.000 | 338.197 | 570.933 |
| H2 | 2.629 | 1.378 | 15.460 | 51.921 | 29.817 | 16.249 | 194.332 | 17.467 | 363.489 | 539.822 |
| H3 | 2.639 | 1.431 | 15.164 | 49.736 | 28.883 | 16.213 | 176.047 | 16.089 | 353.704 | 501.800 |
| H4 | 2.588 | 1.416 | 16.007 | 48.634 | 28.249 | 16.820 | 180.913 | 15.067 | 344.326 | 516.225 |
| H5 | 2.544 | 1.324 | 15.647 | 49.376 | 28.672 | 16.743 | 180.021 | 16.000 | 331.611 | 545.222 |
| H6 | 2.582 | 1.426 | 15.813 | 51.910 | 30.162 | 16.658 | 194.650 | 16.711 | 362.051 | 536.600 |
| H7 | 2.363 | 1.284 | 14.862 | 48.994 | 29.350 | 15.656 | 160.334 | 16.356 | 329.403 | 490.578 |
| H8 | 2.250 | 1.134 | 14.513 | 47.877 | 28.679 | 15.529 | 150.167 | 16.044 | 316.590 | 475.289 |
| H9 | 2.383 | 1.267 | 14.722 | 47.326 | 28.534 | 15.729 | 146.326 | 15.333 | 316.197 | 462.178 |
As seguintes funções *_by() estão disponíveis para calcular as principais estatísticas descritivas por níveis de um fator.
cv_by()Para calcular o coeficiente de variação.max_by()Para calcular os valores máximos.means_by()Para calcular a média aritmética.min_by()Para calcular os valores mínimos.n_by()Para obter o número de observaçõessd_by()Para calcular o desvio padrão amostral.sem_by()Para calcular o erro padrão da média.
Funções úteis
Outras funções úteis também são implementadas. Todos eles funcionam naturalmente com %>%, lidam com dados agrupados com dplyr::group_by() e múltiplas variáveis (todas as variáveis numéricas de .data por padrão).
av_dev()calcula o desvio absoluto médio.ci_mean()calcula o intervalo de confiança para a média.cv()calcula o coeficiente de variação.freq_table()Calcula uma tabela de frequência.hm_mean(),gm_mean()calcula as médias harmônicas e geométricas, respectivamente. A média harmônica é o recíproco da média aritmética dos recíprocos. A média geométrica é a enésima raiz de n produtos.kurt()calcula a curtose como usado em SAS e SPSS.range_data()Calcula o intervalo dos valores.sd_amo(),sd_pop()Calcula a amostra e o desvio padrão populacional, respectivamente.sem()calcula o erro padrão da média.skew()calcula o skewness (assimetria) como usado em SAS e SPSS.sum_dev()calcula a soma dos desvios absolutos.sum_sq_dev()calcula a soma dos desvios quadrados.var_amo(),var_pop()calcula a amostra e a variância populacional.valid_n()Retorna o comprimento válido (nãoNA).
Vamos mostrar alguns exemplos. Observe que
select helpers pode ser usado para selecionar variáveis com base em seus nomes/tipos. O pacote metan apresenta um conjunto de select helpers que pode ser visto com ?metan::Select_helper
# Erro padrão da média para variáveis numéricas que contém (SAB)
df %>% sem(contains("SAB")) %>% print_tbl()
| COMP_SAB | DIAM_SAB |
|---|---|
| 0.232 | 0.109 |
# Intervalo de confiança 0,95 para a média
# Variáveis com largura de nome maior que 3 caracteres
# Agrupado por níveis de ENV
df %>%
group_by(ENV) %>%
ci_mean(width_greater_than(3)) %>%
print_tbl()
## Warning: NA values removed to compute the function. Use 'na.rm = TRUE' to
## suppress this warning.
## To remove rows with NA use `remove_rows_na()'.
## To remove columns with NA use `remove_cols_na()'.
| ENV | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | NFIL |
|---|---|---|---|---|---|---|---|
| AMB1 | 0.042 | 0.043 | 0.405 | 0.643 | 0.769 | 0.366 | 0.688 |
| AMB2 | 0.129 | 0.113 | 0.442 | 0.882 | 0.819 | 0.436 | 0.467 |
| AMB3 | 0.072 | 0.062 | 0.326 | 0.897 | 0.781 | 0.307 | 0.498 |
A função wrapper desc_stat()
Para calcular todas as estatísticas de uma vez, podemos usar desc_stat(). Esta é uma função wrapper em torno das anteriores e pode ser usada para calcular medidas de tendência central, posição e dispersão. Por padrão(stats = "main"), sete estatísticas (coeficiente de variação, máximo, média, mediana, mínimo, desvio padrão da amostra, erro padrão e intervalo de confiança da média) são calculados. Outros valores permitidos são "all" para mostrar todas as estatísticas, "robust" para mostrar estatísticas robustas, e "quantile" para mostrar estatísticas de quantis ou escolher uma (ou mais) estatísticas usando um vetor separado por vírgulas com os nomes das estatísticas, por exemplo, stats = c("mean, cv"). Também podemos usar hist = TRUE para criar um histograma para cada variável.
Todas as estatísticas para todas as variáveis numéricas
all <- desc_stat(df, stats = "all")
## Warning: NA values removed to compute the function. Use 'na.rm = TRUE' to
## suppress this warning.
print_tbl(all)
| variable | av.dev | ci | cv | gmean | hmean | iqr | kurt | mad | max | mean | median | min | n | n.valid | n.missing | n.unique | ps | q2.5 | q25 | q75 | q97.5 | range | sd.amo | sd.pop | se | skew | sum | sum.dev | sum.sq.dev | var.amo | var.pop |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ALT_ESP | 0.285 | 0.058 | 23.858 | 1.275 | 1.236 | 0.552 | -1.381 | 0.415 | 1.88 | 1.313 | 1.340 | 0.75 | 114 | 114 | 0 | 67 | 0.409 | 0.827 | 1.035 | 1.587 | 1.822 | 1.13 | 0.313 | 0.312 | 0.029 | -0.002 | 149.74 | 32.513 | 11.098 | 0.098 | 0.097 |
| ALT_PLANT | 0.348 | 0.070 | 15.236 | 2.416 | 2.406 | 0.680 | -1.505 | 0.534 | 3.04 | 2.463 | 2.520 | 1.71 | 114 | 113 | 1 | 70 | 0.504 | 1.898 | 2.110 | 2.790 | 3.022 | 1.33 | 0.375 | 0.374 | 0.035 | -0.033 | 278.37 | 39.373 | 15.779 | 0.141 | 0.140 |
| COMP_SAB | 2.163 | 0.460 | 8.570 | 28.795 | 28.690 | 4.075 | -0.897 | 2.809 | 34.66 | 28.901 | 28.250 | 23.49 | 114 | 114 | 0 | 104 | 3.018 | 24.477 | 27.020 | 31.095 | 33.004 | 11.17 | 2.477 | 2.466 | 0.232 | 0.037 | 3294.72 | 246.590 | 693.274 | 6.135 | 6.081 |
| COMPES | 1.013 | 0.232 | 8.254 | 15.116 | 15.063 | 1.735 | -0.436 | 1.290 | 17.94 | 15.168 | 15.100 | 12.24 | 114 | 114 | 0 | 91 | 1.285 | 12.632 | 14.365 | 16.100 | 17.500 | 5.70 | 1.252 | 1.246 | 0.117 | -0.129 | 1729.09 | 115.530 | 177.111 | 1.567 | 1.554 |
| DIAM_SAB | 0.963 | 0.216 | 7.270 | 15.984 | 15.941 | 1.815 | -0.605 | 1.319 | 18.28 | 16.026 | 16.030 | 13.28 | 114 | 114 | 0 | 97 | 1.344 | 13.626 | 15.180 | 16.995 | 18.054 | 5.00 | 1.165 | 1.160 | 0.109 | -0.166 | 1826.98 | 109.720 | 153.374 | 1.357 | 1.345 |
| DIAMES | 2.505 | 0.549 | 5.992 | 49.273 | 49.183 | 4.638 | -0.961 | 3.514 | 54.86 | 49.362 | 49.840 | 43.48 | 114 | 114 | 0 | 107 | 3.435 | 43.880 | 47.090 | 51.728 | 54.009 | 11.38 | 2.958 | 2.945 | 0.277 | -0.248 | 5627.22 | 285.541 | 988.680 | 8.749 | 8.673 |
| MGE | 29.077 | 6.405 | 20.218 | 167.159 | 163.481 | 55.565 | -0.884 | 39.815 | 250.89 | 170.743 | 171.735 | 105.72 | 114 | 114 | 0 | 113 | 41.159 | 112.500 | 141.970 | 197.535 | 229.725 | 145.17 | 34.520 | 34.369 | 3.233 | -0.014 | 19464.66 | 3314.735 | 134658.072 | 1191.664 | 1181.211 |
| MMG | 39.450 | 9.305 | 14.918 | 332.332 | 328.354 | 65.100 | -0.266 | 46.524 | 451.68 | 336.168 | 336.050 | 226.60 | 114 | 114 | 0 | 114 | 48.222 | 232.476 | 305.905 | 371.005 | 430.760 | 225.08 | 50.149 | 49.929 | 4.697 | -0.089 | 38323.16 | 4497.340 | 284189.109 | 2514.948 | 2492.887 |
| NFIL | 1.391 | 0.325 | 10.833 | 16.062 | 15.971 | 2.400 | 0.148 | 1.779 | 21.20 | 16.154 | 16.000 | 12.40 | 114 | 114 | 0 | 20 | 1.778 | 13.200 | 14.800 | 17.200 | 20.070 | 8.80 | 1.750 | 1.742 | 0.164 | 0.448 | 1841.60 | 158.526 | 346.083 | 3.063 | 3.036 |
| NGE | 55.433 | 13.616 | 14.314 | 450.933 | 497.885 | 73.350 | 0.172 | 55.894 | 696.60 | 508.018 | 504.400 | 354.00 | 114 | 112 | 2 | 107 | 54.333 | 381.230 | 465.500 | 538.850 | 669.285 | 342.60 | 72.718 | 72.392 | 6.871 | 0.410 | 56898.00 | 6208.471 | 586955.724 | 5287.889 | 5240.676 |
Crie um histograma para cada variável
stat1 <-
df %>%
desc_stat(ALT_ESP, ALT_PLANT, COMP_SAB,
hist = TRUE)
## Warning: NA values removed to compute the function. Use 'na.rm = TRUE' to
## suppress this warning.
## Warning: Removed 1 rows containing non-finite values (stat_bin).
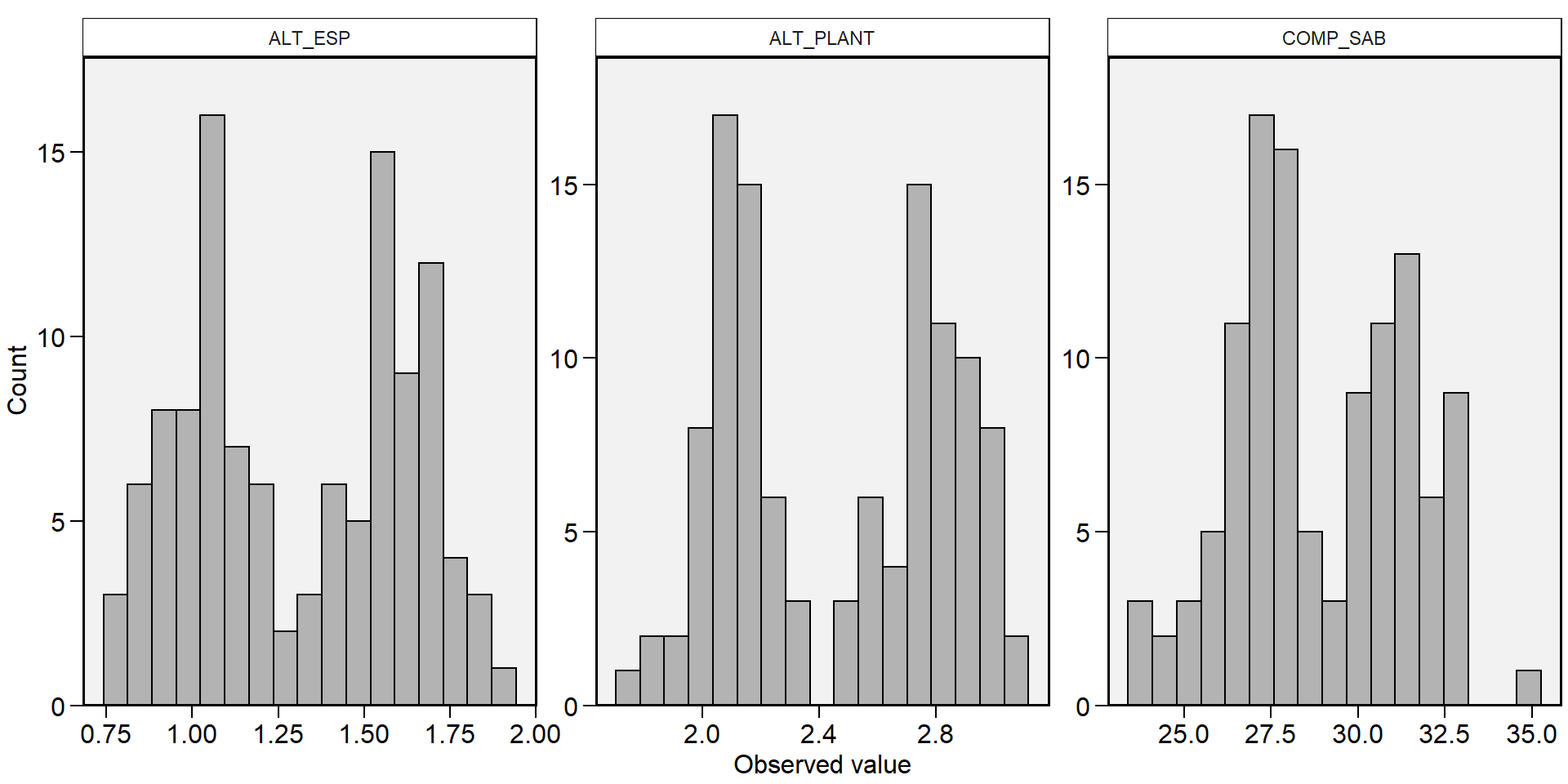
print_tbl(stat1)
| variable | cv | max | mean | median | min | sd.amo | se | ci |
|---|---|---|---|---|---|---|---|---|
| ALT_ESP | 23.858 | 1.88 | 1.313 | 1.34 | 0.75 | 0.313 | 0.029 | 0.058 |
| ALT_PLANT | 15.236 | 3.04 | 2.463 | 2.52 | 1.71 | 0.375 | 0.035 | 0.070 |
| COMP_SAB | 8.570 | 34.66 | 28.901 | 28.25 | 23.49 | 2.477 | 0.232 | 0.460 |
Estatísticas por níveis de fatores
Para calcular as estatísticas para cada nível de um fator, use o argumento by. Além disso, é possível selecionar as estatísticas a serem computadas usando o argumento stats, que aceita um único nome de estatística, por exemplo,"mean", ou um vetor de nomes separados por vírgula com " no início e apenas no final do vetor. Observe que os nomes das estatísticas NÃO diferenciam maiúsculas de minúsculas, ou seja, "mean", "Mean" ou "MEAN" são reconhecidos. Vírgulas ou espaços podem ser usados separar os nomes das estatísticas.
- Todas as opções abaixo funcionarão:
stats = c("mean, se, cv, max, min")stats = c("mean se cv max min")stats = c("MEAN, Se, CV max Min")
stats_c <-
desc_stat(df,
contains("C"),
stats = ("mean, se, cv, max, min, n, n.valid"),
by = ENV)
print_tbl(stats_c)
| ENV | variable | mean | se | cv | max | min | n | n.valid |
|---|---|---|---|---|---|---|---|---|
| AMB1 | COMP_SAB | 29.866 | 0.379 | 7.610 | 34.66 | 25.93 | 36 | 36 |
| AMB1 | COMPES | 15.637 | 0.200 | 7.656 | 17.50 | 12.24 | 36 | 36 |
| AMB2 | COMP_SAB | 28.464 | 0.405 | 8.880 | 33.02 | 23.85 | 39 | 39 |
| AMB2 | COMPES | 15.233 | 0.218 | 8.954 | 17.94 | 12.50 | 39 | 39 |
| AMB3 | COMP_SAB | 28.448 | 0.386 | 8.472 | 33.16 | 23.49 | 39 | 39 |
| AMB3 | COMPES | 14.668 | 0.161 | 6.853 | 16.84 | 12.24 | 39 | 39 |
Podemos converter os resultados acima em um formato wide usando a função desc_wider()
desc_wider(stats_c, mean) %>% print_tbl()
| ENV | COMP_SAB | COMPES |
|---|---|---|
| AMB1 | 29.866 | 15.637 |
| AMB2 | 28.464 | 15.233 |
| AMB3 | 28.448 | 14.668 |
Para calcular as estatísticas descritivas por mais de uma variável de agrupamento, é necessário passar os dados agrupados para o argumento .data com a função group_by(). Vamos calcular a média, o erro padrão da média e o tamanho da amostra para as variáveis MGE e MMG para todas as combinações dos fatores ENV e GEN.
stats_grp <-
df %>%
group_by(ENV, GEN) %>%
desc_stat(MGE, MMG,
stats = c("mean, se, n"))
print_tbl(stats_grp)
| ENV | GEN | variable | mean | se | n |
|---|---|---|---|---|---|
| AMB1 | H10 | MGE | 192.410 | 8.880 | 3 |
| AMB1 | H10 | MMG | 373.507 | 13.949 | 3 |
| AMB1 | H11 | MGE | 188.253 | 15.158 | 3 |
| AMB1 | H11 | MMG | 353.037 | 16.286 | 3 |
| AMB1 | H12 | MGE | 180.483 | 9.840 | 3 |
| AMB1 | H12 | MMG | 332.843 | 6.059 | 3 |
| AMB1 | H13 | MGE | 218.603 | 10.153 | 3 |
| AMB1 | H13 | MMG | 368.107 | 26.883 | 3 |
| AMB1 | H2 | MGE | 203.833 | 4.324 | 3 |
| AMB1 | H2 | MMG | 328.863 | 26.630 | 3 |
| AMB1 | H3 | MGE | 198.210 | 5.800 | 3 |
| AMB1 | H3 | MMG | 325.013 | 10.540 | 3 |
| AMB1 | H4 | MGE | 201.823 | 7.017 | 3 |
| AMB1 | H4 | MMG | 337.593 | 16.263 | 3 |
| AMB1 | H5 | MGE | 192.700 | 5.941 | 3 |
| AMB1 | H5 | MMG | 356.493 | 9.010 | 3 |
| AMB1 | H6 | MGE | 231.853 | 9.689 | 3 |
| AMB1 | H6 | MMG | 412.083 | 18.070 | 3 |
| AMB1 | H7 | MGE | 181.547 | 12.826 | 3 |
| AMB1 | H7 | MMG | 338.097 | 3.564 | 3 |
| AMB1 | H8 | MGE | 196.110 | 7.991 | 3 |
| AMB1 | H8 | MMG | 365.723 | 28.822 | 3 |
| AMB1 | H9 | MGE | 204.173 | 11.909 | 3 |
| AMB1 | H9 | MMG | 413.087 | 14.194 | 3 |
| AMB2 | H1 | MGE | 188.197 | 5.907 | 3 |
| AMB2 | H1 | MMG | 388.933 | 0.984 | 3 |
| AMB2 | H10 | MGE | 159.897 | 8.618 | 3 |
| AMB2 | H10 | MMG | 315.937 | 14.960 | 3 |
| AMB2 | H11 | MGE | 163.607 | 5.068 | 3 |
| AMB2 | H11 | MMG | 341.970 | 10.151 | 3 |
| AMB2 | H12 | MGE | 131.273 | 4.983 | 3 |
| AMB2 | H12 | MMG | 313.167 | 35.943 | 3 |
| AMB2 | H13 | MGE | 169.070 | 11.196 | 3 |
| AMB2 | H13 | MMG | 326.857 | 2.293 | 3 |
| AMB2 | H2 | MGE | 218.857 | 2.571 | 3 |
| AMB2 | H2 | MMG | 415.673 | 7.743 | 3 |
| AMB2 | H3 | MGE | 190.943 | 18.716 | 3 |
| AMB2 | H3 | MMG | 400.043 | 26.337 | 3 |
| AMB2 | H4 | MGE | 197.463 | 4.510 | 3 |
| AMB2 | H4 | MMG | 388.657 | 10.715 | 3 |
| AMB2 | H5 | MGE | 186.243 | 12.477 | 3 |
| AMB2 | H5 | MMG | 332.840 | 13.274 | 3 |
| AMB2 | H6 | MGE | 215.030 | 12.091 | 3 |
| AMB2 | H6 | MMG | 346.030 | 8.796 | 3 |
| AMB2 | H7 | MGE | 143.800 | 4.810 | 3 |
| AMB2 | H7 | MMG | 292.070 | 3.821 | 3 |
| AMB2 | H8 | MGE | 112.960 | 3.405 | 3 |
| AMB2 | H8 | MMG | 236.283 | 5.929 | 3 |
| AMB2 | H9 | MGE | 112.333 | 5.182 | 3 |
| AMB2 | H9 | MMG | 241.130 | 9.561 | 3 |
| AMB3 | H1 | MGE | 156.817 | 13.734 | 3 |
| AMB3 | H1 | MMG | 368.770 | 26.866 | 3 |
| AMB3 | H10 | MGE | 120.557 | 3.643 | 3 |
| AMB3 | H10 | MMG | 247.260 | 7.641 | 3 |
| AMB3 | H11 | MGE | 140.717 | 15.513 | 3 |
| AMB3 | H11 | MMG | 288.630 | 14.700 | 3 |
| AMB3 | H12 | MGE | 148.363 | 4.707 | 3 |
| AMB3 | H12 | MMG | 283.630 | 4.198 | 3 |
| AMB3 | H13 | MGE | 181.603 | 8.788 | 3 |
| AMB3 | H13 | MMG | 319.627 | 35.063 | 3 |
| AMB3 | H2 | MGE | 160.307 | 3.045 | 3 |
| AMB3 | H2 | MMG | 345.930 | 10.698 | 3 |
| AMB3 | H3 | MGE | 138.987 | 9.807 | 3 |
| AMB3 | H3 | MMG | 336.057 | 7.297 | 3 |
| AMB3 | H4 | MGE | 143.453 | 8.352 | 3 |
| AMB3 | H4 | MMG | 306.727 | 23.382 | 3 |
| AMB3 | H5 | MGE | 161.120 | 14.097 | 3 |
| AMB3 | H5 | MMG | 305.500 | 7.953 | 3 |
| AMB3 | H6 | MGE | 137.067 | 9.840 | 3 |
| AMB3 | H6 | MMG | 328.040 | 23.076 | 3 |
| AMB3 | H7 | MGE | 155.657 | 11.222 | 3 |
| AMB3 | H7 | MMG | 358.043 | 3.251 | 3 |
| AMB3 | H8 | MGE | 141.430 | 12.736 | 3 |
| AMB3 | H8 | MMG | 347.763 | 22.006 | 3 |
| AMB3 | H9 | MGE | 122.470 | 5.238 | 3 |
| AMB3 | H9 | MMG | 294.373 | 20.413 | 3 |