Índices de estabilidade
library(metan)
## Registered S3 method overwritten by 'GGally':
## method from
## +.gg ggplot2
## |=========================================================|
## | Multi-Environment Trial Analysis (metan) v1.15.0 |
## | Author: Tiago Olivoto |
## | Type 'citation('metan')' to know how to cite metan |
## | Type 'vignette('metan_start')' for a short tutorial |
## | Visit 'https://bit.ly/pkgmetan' for a complete tutorial |
## |=========================================================|
library(rio)
df_ge <- import("http://bit.ly/df_ge", setclass = "tbl")
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
Desempenho dos genótipos em cada ambiente
A função ge_plot() pode ser usada para visualizar o desempenho do genótipo nos ambientes.
a <- ge_plot(df_ge, ENV, GEN, MMG)
b <- ge_plot(df_ge, ENV, GEN, MMG, type = 2)
arrange_ggplot(a, b, tag_levels = "a")
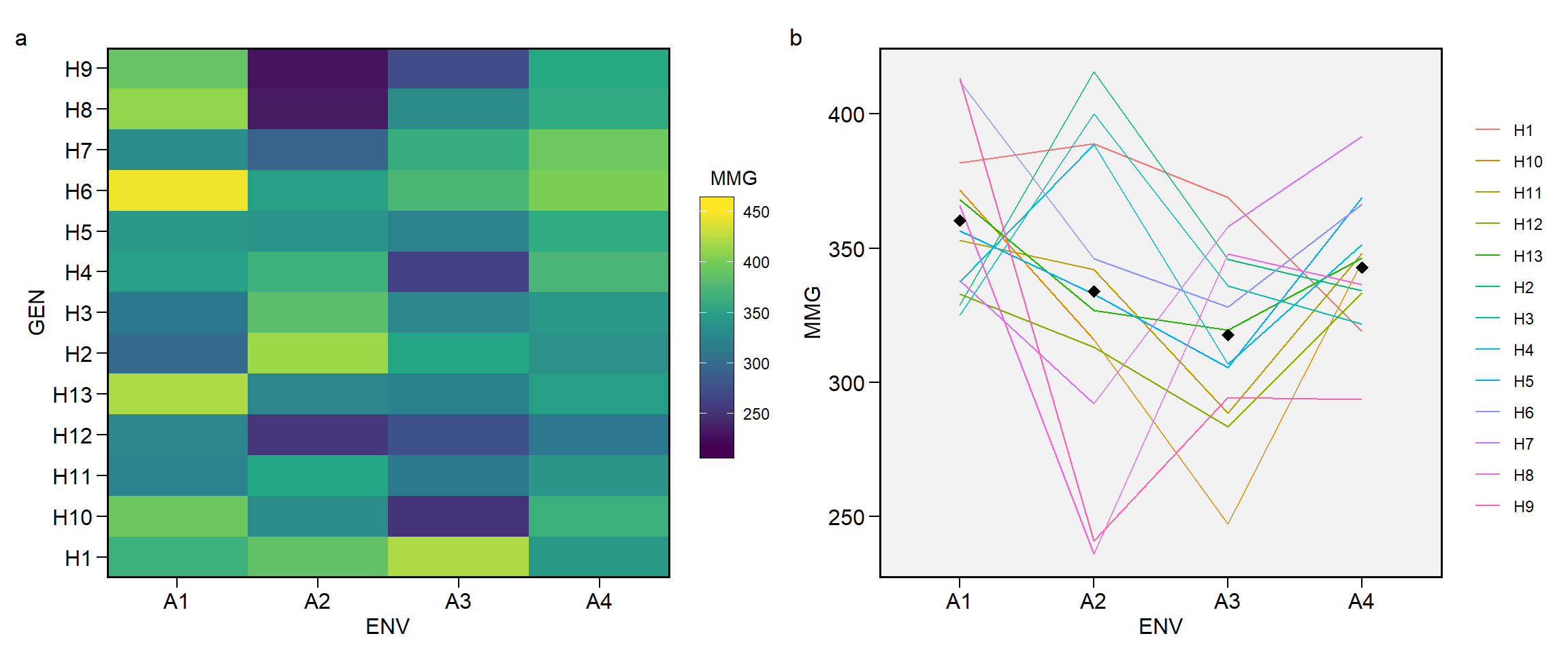
Para identificar o genótipo ganhador em cada ambiente, podemos usar a função ge_winners().
ge_winners(df_ge, ENV, GEN, resp = everything()) %>% print_tbl()
| ENV | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| A1 | H3 | H1 | H6 | H6 | H8 | H6 | H6 | H2 | H9 | H2 |
| A2 | H2 | H1 | H6 | H2 | H2 | H6 | H2 | H2 | H2 | H6 |
| A3 | H13 | H13 | H4 | H13 | H6 | H2 | H13 | H13 | H1 | H13 |
| A4 | H5 | H5 | H7 | H11 | H5 | H7 | H7 | H11 | H7 | H9 |
Ou obter a classificação dos genótipos em cada ambiente.
winners <-
ge_winners(df_ge, ENV, GEN,
resp = everything(),
type = "ranks",
better = c("l, l, h, h, h, h, h, h, h, h"))
print_tbl(winners)
| ENV | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| A1 | H8 | H2 | H6 | H6 | H8 | H6 | H6 | H2 | H9 | H2 |
| A1 | H12 | H8 | H11 | H13 | H9 | H11 | H13 | H13 | H6 | H13 |
| A1 | H1 | H12 | H10 | H10 | H6 | H9 | H9 | H3 | H1 | H3 |
| A1 | H11 | H6 | H4 | H9 | H10 | H10 | H2 | H7 | H10 | H4 |
| A1 | H6 | H11 | H5 | H8 | H13 | H5 | H1 | H12 | H13 | H6 |
| A1 | H13 | H13 | H9 | H2 | H7 | H4 | H4 | H8 | H8 | H12 |
| A1 | H7 | H3 | H3 | H3 | H12 | H8 | H3 | H6 | H5 | H7 |
| A1 | H10 | H5 | H7 | H1 | H11 | H3 | H8 | H10 | H11 | H8 |
| A1 | H2 | H7 | H1 | H7 | H5 | H7 | H5 | H4 | H7 | H5 |
| A1 | H5 | H10 | H12 | H4 | H1 | H1 | H10 | H1 | H4 | H11 |
| A1 | H4 | H9 | H8 | H12 | H3 | H12 | H11 | H11 | H12 | H1 |
| A1 | H9 | H4 | H2 | H5 | H4 | H2 | H7 | H9 | H2 | H10 |
| A1 | H3 | H1 | H13 | H11 | H2 | H13 | H12 | H5 | H3 | H9 |
| A2 | H8 | H8 | H6 | H2 | H2 | H6 | H2 | H2 | H2 | H6 |
| A2 | H10 | H12 | H4 | H1 | H13 | H4 | H6 | H1 | H3 | H5 |
| A2 | H12 | H10 | H5 | H6 | H1 | H5 | H4 | H6 | H1 | H2 |
| A2 | H7 | H11 | H2 | H3 | H3 | H13 | H3 | H13 | H4 | H13 |
| A2 | H11 | H13 | H13 | H13 | H4 | H2 | H1 | H9 | H6 | H10 |
| A2 | H9 | H7 | H10 | H4 | H5 | H10 | H5 | H8 | H11 | H4 |
| A2 | H13 | H9 | H3 | H5 | H11 | H3 | H13 | H5 | H5 | H7 |
| A2 | H5 | H5 | H11 | H7 | H12 | H11 | H11 | H7 | H13 | H1 |
| A2 | H6 | H6 | H1 | H11 | H7 | H7 | H10 | H12 | H10 | H9 |
| A2 | H4 | H4 | H7 | H12 | H6 | H1 | H7 | H3 | H12 | H11 |
| A2 | H1 | H2 | H8 | H10 | H10 | H9 | H12 | H4 | H7 | H8 |
| A2 | H3 | H3 | H9 | H9 | H9 | H8 | H8 | H10 | H9 | H3 |
| A2 | H2 | H1 | H12 | H8 | H8 | H12 | H9 | H11 | H8 | H12 |
| A3 | H9 | H9 | H4 | H13 | H6 | H2 | H13 | H13 | H1 | H13 |
| A3 | H10 | H4 | H2 | H2 | H1 | H4 | H5 | H5 | H7 | H5 |
| A3 | H3 | H3 | H13 | H1 | H5 | H5 | H2 | H6 | H8 | H12 |
| A3 | H4 | H5 | H9 | H5 | H7 | H3 | H1 | H11 | H2 | H10 |
| A3 | H5 | H10 | H1 | H6 | H2 | H13 | H7 | H12 | H3 | H11 |
| A3 | H8 | H2 | H3 | H7 | H8 | H9 | H12 | H1 | H6 | H4 |
| A3 | H11 | H8 | H8 | H12 | H3 | H8 | H4 | H2 | H13 | H2 |
| A3 | H6 | H1 | H5 | H3 | H4 | H1 | H8 | H7 | H4 | H7 |
| A3 | H2 | H11 | H7 | H11 | H13 | H11 | H11 | H10 | H5 | H1 |
| A3 | H7 | H7 | H11 | H8 | H9 | H7 | H3 | H3 | H9 | H3 |
| A3 | H1 | H6 | H10 | H4 | H11 | H10 | H6 | H8 | H11 | H9 |
| A3 | H12 | H12 | H12 | H10 | H12 | H6 | H9 | H9 | H12 | H6 |
| A3 | H13 | H13 | H6 | H9 | H10 | H12 | H10 | H4 | H10 | H8 |
| A4 | H9 | H9 | H7 | H11 | H5 | H7 | H7 | H11 | H7 | H9 |
| A4 | H10 | H3 | H8 | H5 | H10 | H8 | H5 | H1 | H5 | H1 |
| A4 | H3 | H11 | H9 | H1 | H6 | H9 | H4 | H12 | H6 | H8 |
| A4 | H6 | H2 | H6 | H4 | H11 | H6 | H8 | H4 | H4 | H4 |
| A4 | H2 | H6 | H5 | H7 | H12 | H5 | H1 | H5 | H11 | H5 |
| A4 | H7 | H12 | H1 | H6 | H7 | H1 | H10 | H10 | H13 | H7 |
| A4 | H12 | H13 | H2 | H10 | H1 | H11 | H11 | H9 | H10 | H12 |
| A4 | H8 | H8 | H4 | H12 | H4 | H4 | H9 | H13 | H8 | H11 |
| A4 | H4 | H10 | H10 | H8 | H8 | H2 | H12 | H7 | H2 | H10 |
| A4 | H13 | H7 | H11 | H3 | H9 | H10 | H6 | H8 | H12 | H2 |
| A4 | H11 | H1 | H12 | H9 | H2 | H13 | H2 | H6 | H3 | H3 |
| A4 | H1 | H4 | H13 | H2 | H3 | H12 | H13 | H3 | H1 | H6 |
| A4 | H5 | H5 | H3 | H13 | H13 | H3 | H3 | H2 | H9 | H13 |
Para mais detalhes sobre os testes, podemos usar ge_details()
details <- ge_details(df_ge, ENV, GEN, resp = everything())
print_tbl(details)
| Parameters | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| Mean | 2.48 | 1.34 | 15.16 | 49.54 | 29.01 | 15.97 | 172.94 | 16.12 | 338.67 | 511.64 |
| SE | 0.03 | 0.02 | 0.1 | 0.22 | 0.18 | 0.09 | 2.62 | 0.13 | 3.77 | 5.82 |
| SD | 0.33 | 0.28 | 1.25 | 2.76 | 2.3 | 1.17 | 32.67 | 1.63 | 46.91 | 72.41 |
| CV | 13.44 | 21.16 | 8.28 | 5.58 | 7.95 | 7.34 | 18.95 | 10.16 | 13.9 | 14.2 |
| Min | 1.71 (H11 in A3) | 0.75 (H8 in A2) | 11.5 (H3 in A4) | 43.48 (H9 in A3) | 23.49 (H10 in A3) | 12.9 (H3 in A4) | 105.72 (H9 in A2) | 12.4 (H4 in A3) | 218.32 (H9 in A4) | 331.8 (H13 in A4) |
| Max | 3.04 (H3 in A1) | 1.88 (H1 in A2) | 17.94 (H6 in A2) | 54.86 (H6 in A1) | 34.66 (H8 in A1) | 18.56 (H7 in A4) | 250.89 (H6 in A1) | 21.2 (H2 in A1) | 451.68 (H3 in A2) | 696.6 (H2 in A1) |
| MinENV | A3 (2.17) | A3 (1.08) | A3 (14.67) | A3 (47.87) | A3 (28.45) | A3 (15.77) | A3 (146.81) | A3 (15.78) | A3 (317.72) | A3 (467.9) |
| MaxENV | A1 (2.79) | A1 (1.58) | A1 (15.62) | A1 (51.61) | A1 (29.73) | A1 (16.4) | A1 (199.44) | A1 (16.89) | A1 (360.34) | A1 (557.87) |
| MinGEN | H10 (2.31) | H8 (1.21) | H12 (14.28) | H9 (47.59) | H12 (28.24) | H12 (14.81) | H9 (152.75) | H9 (15.47) | H9 (310.57) | H3 (491.23) |
| MaxGEN | H1 (2.62) | H1 (1.5) | H6 (15.76) | H6 (51.53) | H6 (30.25) | H5 (16.59) | H6 (188) | H13 (17.43) | H1 (364.63) | H5 (541.7) |
Matriz dupla entrada
A função make_mat() pode ser usada para produzir uma tabela bidirecional com as médias genótipo-ambiente.
mat <- make_mat(df_ge, GEN, ENV, MMG)
print_tbl(mat)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| H1 | 381.821 | 388.932 | 368.768 | 318.987 |
| H10 | 371.627 | 315.936 | 247.261 | 345.017 |
| H11 | 353.036 | 341.971 | 288.631 | 348.023 |
| H12 | 332.847 | 313.167 | 283.633 | 333.544 |
| H13 | 368.108 | 326.857 | 319.627 | 346.361 |
| H2 | 328.861 | 415.675 | 345.927 | 334.220 |
| H3 | 325.013 | 400.045 | 336.053 | 321.801 |
| H4 | 337.590 | 388.658 | 306.729 | 351.451 |
| H5 | 356.492 | 332.841 | 305.497 | 368.845 |
| H6 | 412.084 | 346.030 | 328.038 | 366.269 |
| H7 | 338.093 | 292.069 | 358.046 | 391.762 |
| H8 | 365.720 | 236.281 | 347.763 | 336.371 |
| H9 | 413.088 | 241.127 | 294.359 | 293.694 |
Efeitos de interação genótipo-ambiente
A função ge_effects() é usada para calcular os efeitos da interação genótipo-ambiente.
ge_ef <- ge_effects(df_ge, ENV, GEN, MMG)
print_tbl(ge_ef$MMG)
| GEN | A1 | A2 | A3 | A4 |
|---|---|---|---|---|
| H1 | -4.477 | 29.157 | 25.089 | -49.769 |
| H10 | 29.996 | 0.828 | -51.751 | 20.927 |
| H11 | -1.550 | 13.908 | -23.336 | 10.978 |
| H12 | -4.621 | 2.221 | -11.216 | 13.617 |
| H13 | 6.199 | -8.530 | 0.337 | 1.993 |
| H2 | -48.980 | 64.356 | 10.705 | -26.080 |
| H3 | -42.386 | 59.169 | 11.274 | -28.057 |
| H4 | -30.187 | 47.402 | -18.430 | 1.215 |
| H5 | -6.097 | -3.226 | -14.473 | 23.797 |
| H6 | 27.308 | -12.224 | -14.118 | -0.966 |
| H7 | -28.570 | -48.071 | 34.002 | 42.640 |
| H8 | 22.515 | -80.401 | 47.177 | 10.708 |
| H9 | 80.850 | -64.588 | 4.740 | -21.002 |
# o mesmo efeito é calculado com o resíduo do modelo aditivo
ge_ef2 <-
df_ge %>%
means_by(GEN, ENV) %>%
lm(MMG ~ GEN + ENV, data = .) %>%
residuals() %>%
matrix(nrow = 13, byrow = TRUE)
print_tbl(ge_ef2)
| -4.477 | 29.157 | 25.089 | -49.769 |
| 29.996 | 0.828 | -51.751 | 20.927 |
| -1.550 | 13.908 | -23.336 | 10.978 |
| -4.621 | 2.221 | -11.216 | 13.617 |
| 6.199 | -8.530 | 0.337 | 1.993 |
| -48.980 | 64.356 | 10.705 | -26.080 |
| -42.386 | 59.169 | 11.274 | -28.057 |
| -30.187 | 47.402 | -18.430 | 1.215 |
| -6.097 | -3.226 | -14.473 | 23.797 |
| 27.308 | -12.224 | -14.118 | -0.966 |
| -28.570 | -48.071 | 34.002 | 42.640 |
| 22.515 | -80.401 | 47.177 | 10.708 |
| 80.850 | -64.588 | 4.740 | -21.002 |
Genótipo + interação genótipo-ambiente (GGE)
Para obter o efeito GGE, usamos argumento type =" gge " na função ge_effects().
gge_ef <- ge_effects(df_ge, ENV, GEN, MMG, type = "gge")
print_tbl(gge_ef$MMG)
| GEN | A1 | A2 | A3 | A4 |
|---|---|---|---|---|
| H1 | 21.484 | 55.117 | 51.050 | -23.809 |
| H10 | 11.290 | -17.878 | -70.457 | 2.221 |
| H11 | -7.301 | 8.156 | -29.087 | 5.227 |
| H12 | -27.489 | -20.647 | -34.085 | -9.251 |
| H13 | 7.771 | -6.958 | 1.909 | 3.565 |
| H2 | -31.476 | 81.861 | 28.209 | -8.576 |
| H3 | -35.324 | 66.231 | 18.336 | -20.995 |
| H4 | -22.747 | 54.843 | -10.989 | 8.656 |
| H5 | -3.845 | -0.974 | -12.221 | 26.049 |
| H6 | 51.747 | 12.215 | 10.320 | 23.473 |
| H7 | -22.244 | -41.745 | 40.328 | 48.966 |
| H8 | 5.383 | -97.533 | 30.045 | -6.425 |
| H9 | 52.751 | -92.687 | -23.359 | -49.101 |
# o mesmo efeito é calculado com o resíduo do modelo aditivo
gge_ef2 <-
df_ge %>%
means_by(GEN, ENV) %>%
lm(MMG ~ ENV, data = .) %>%
residuals() %>%
matrix(nrow = 13, byrow = TRUE)
print_tbl(gge_ef2)
| 21.484 | 55.117 | 51.050 | -23.809 |
| 11.290 | -17.878 | -70.457 | 2.221 |
| -7.301 | 8.156 | -29.087 | 5.227 |
| -27.489 | -20.647 | -34.085 | -9.251 |
| 7.771 | -6.958 | 1.909 | 3.565 |
| -31.476 | 81.861 | 28.209 | -8.576 |
| -35.324 | 66.231 | 18.336 | -20.995 |
| -22.747 | 54.843 | -10.989 | 8.656 |
| -3.845 | -0.974 | -12.221 | 26.049 |
| 51.747 | 12.215 | 10.320 | 23.473 |
| -22.244 | -41.745 | 40.328 | 48.966 |
| 5.383 | -97.533 | 30.045 | -6.425 |
| 52.751 | -92.687 | -23.359 | -49.101 |
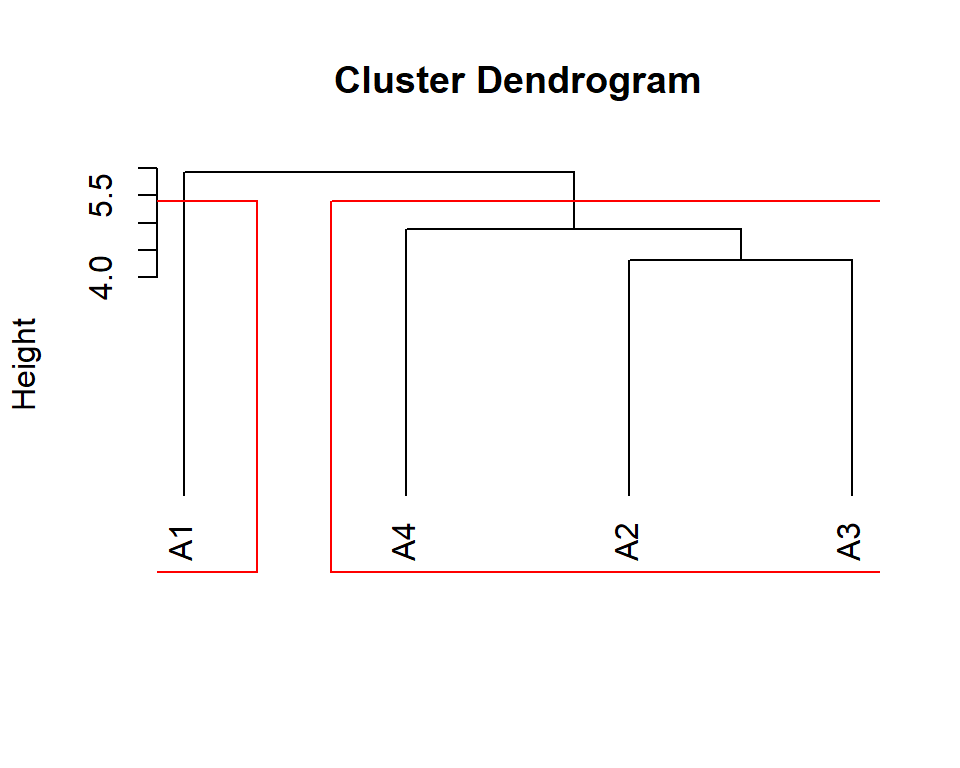
Agrupamento de ambientes
A função ge_cluster() computa uma análise de agrupamento para agrupar ambientes com base em suas semelhanças usando uma distância euclidiana baseada em dados padronizados.
d1 <- ge_cluster(df_ge, ENV, GEN, MMG, nclust = 2)
plot(d1, nclust = 2)
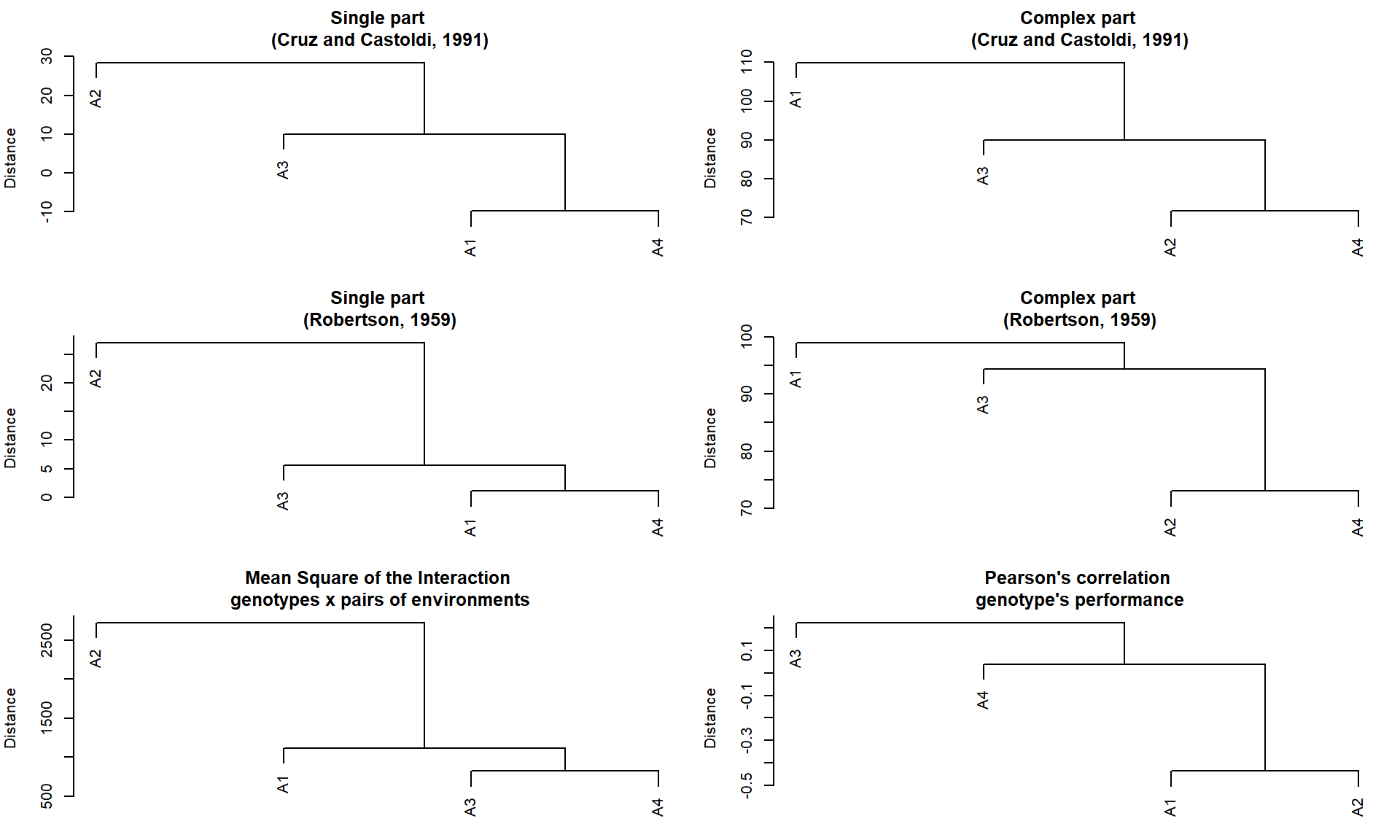
A função env_dissimilarity() calcula a dissimilaridade entre os ambientes de teste usando:
-
A partição da partição do quadrado médio da interação genótipo-ambiente (MS_GE) em partes simples (S) e complexas (C), de acordo com Robertson (1959)1, onde \(S = \frac{1}{2} (\sqrt {Q_1} - \sqrt{Q_2}) ^ 2)\) e \(C = (1-r) \sqrt {Q1-Q2}\), sendo \(r\) a correlação entre a média do genótipo nos dois ambientes; e \(Q_1\) e \(Q_2\) o quadrado médio do genótipo nos ambientes 1 e 2, respectivamente.
-
A decomposição do MS_GE proposta por Cruz e Castoldi (1991)2 em que a parte complexa é dada por \(C = \sqrt {(1-r) ^ 3 Q1 Q2}\).
-
O quadrado médio da interação entre genótipos e pares de ambientes.
-
Os coeficientes de correlação entre a média dos genótipos em cada par de ambiente.
mod <- env_dissimilarity(df_ge, ENV, GEN, BLOCO, MMG)
## Evaluating trait Y |=============================================| 100% 00:00:00
# Coeficiente de correlação de Pearson
print_tbl(mod$MMG$correlation)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 1.000 | -0.435 | -0.100 | -0.234 |
| A2 | -0.435 | 1.000 | 0.221 | 0.038 |
| A3 | -0.100 | 0.221 | 1.000 | 0.091 |
| A4 | -0.234 | 0.038 | 0.091 | 1.000 |
# Quadrado médio GxEjj '
print_tbl(mod$MMG$MSGE)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 0.000 | 2723.279 | 1116.317 | 901.421 |
| A2 | 2723.279 | 0.000 | 1745.096 | 1837.028 |
| A3 | 1116.317 | 1745.096 | 0.000 | 819.735 |
| A4 | 901.421 | 1837.028 | 819.735 | 0.000 |
#% Parte simples do QM GxEjj '(Robertson, 1959)
print_tbl(mod$MMG$SPART_RO)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 0.000 | 13.476 | 1.203 | 1.073 |
| A2 | 13.476 | 0.000 | 13.753 | 26.991 |
| A3 | 1.203 | 13.753 | 0.000 | 5.600 |
| A4 | 1.073 | 26.991 | 5.600 | 0.000 |
#% Da parte complexa do QM GxEjj '(Robertson, 1959)
print_tbl(mod$MMG$CPART_RO)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 0.000 | 86.524 | 98.797 | 98.927 |
| A2 | 86.524 | 0.000 | 86.247 | 73.009 |
| A3 | 98.797 | 86.247 | 0.000 | 94.400 |
| A4 | 98.927 | 73.009 | 94.400 | 0.000 |
#% Parte simples do QM GxEjj '(Cruz e Castoldi, 1991)
print_tbl(mod$MMG$SPART_CC)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 0.000 | -3.664 | -3.634 | -9.882 |
| A2 | -3.664 | 0.000 | 23.899 | 28.388 |
| A3 | -3.634 | 23.899 | 0.000 | 9.993 |
| A4 | -9.882 | 28.388 | 9.993 | 0.000 |
#% Parte complexa do QM GxEjj '(Cruz e Castoldi, 1991)
print_tbl(mod$MMG$CPART_CC)
| A1 | A2 | A3 | A4 | |
|---|---|---|---|---|
| A1 | 0.000 | 103.664 | 103.634 | 109.882 |
| A2 | 103.664 | 0.000 | 76.101 | 71.612 |
| A3 | 103.634 | 76.101 | 0.000 | 90.007 |
| A4 | 109.882 | 71.612 | 90.007 | 0.000 |
Para obter dendrogramas com base na matriz acima, podemos usar plot(). Os dendrogramas são baseados no algoritmo de agrupamento hierárquico UPGMA (método de grupo de pares não ponderados usando médias aritméticas).
plot(mod)
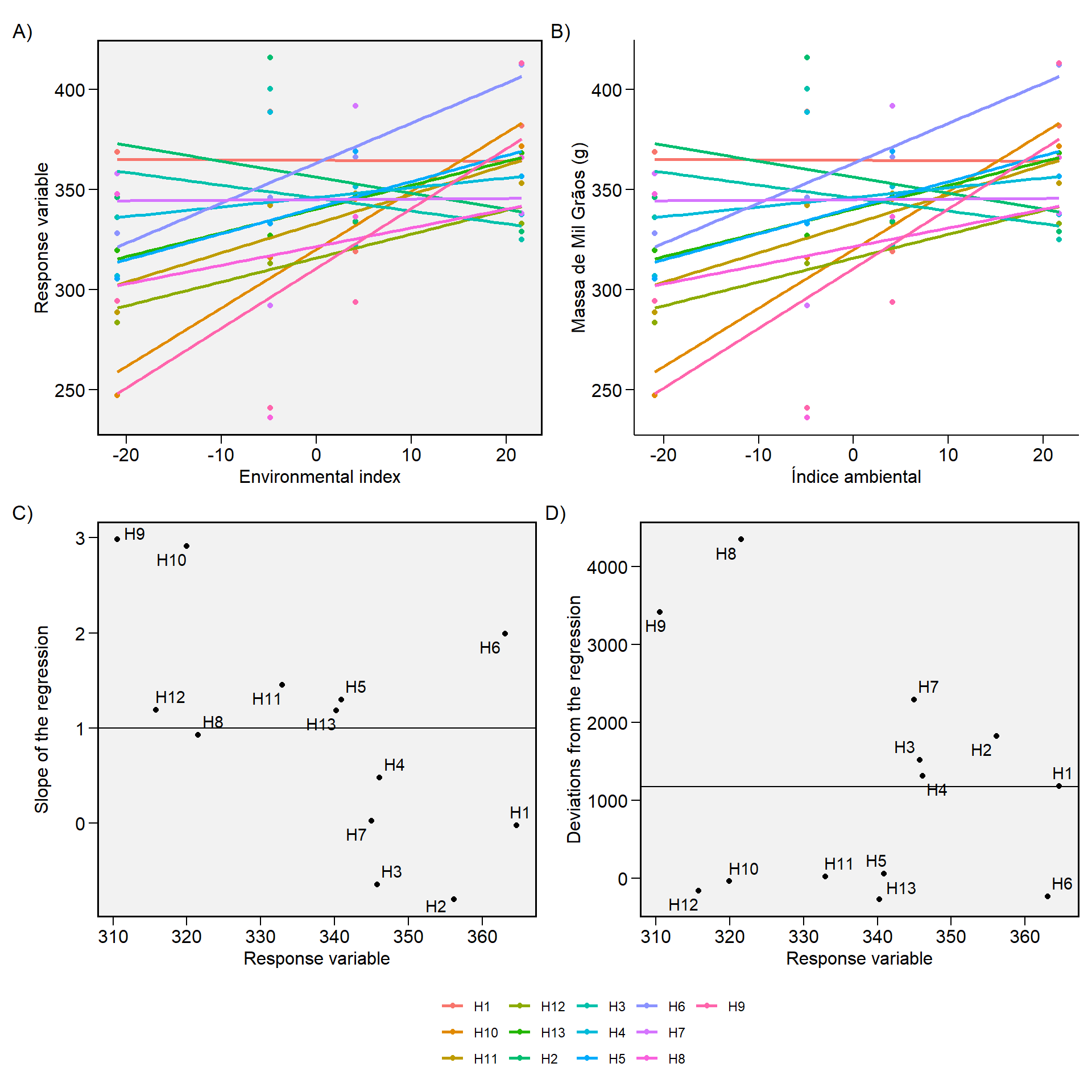
Análise de regressão conjunta
Eberhart e Russell (1966)3 popularizaram a análise de estabilidade baseada em regressão. Neste procedimento, a análise de adaptabilidade e estabilidade é realizada por meio de ajustes de equações de regressão onde a variável dependente é predita em função de um índice ambiental, de acordo com o seguinte modelo:
$$ \mathop Y \nolimits_{ij} = {\beta_{0i}} + {\beta_{1i}}{I_j} + {\delta_{ij}} + {\bar \varepsilon_{ij}} $$
onde \({\beta_{0i}}\) é a média do genótipo \(i\) (\(i\) = 1, 2, …, I); \({\beta_{1i}}\) é a resposta linear (slope) do genótipo \(i\) ao índice ambiental; \(Ij\) é o índice ambiental (\(j\) = 1, 2, …, \(e\)), onde \({I_j} = [(y_{.J} / g) - (y_{..}/ge)]\), \({\delta_{ij}}\) é o desvio da regressão e \({\ bar \varepsilon_{ij}}\) é o erro experimental.
O modelo é ajustado com a função ge_reg(). Os métodos S3 plot() e summary () podem ser usados para explorar o modelo ajustado.
reg_model <- ge_reg(df_ge,
env = ENV,
gen = GEN,
rep = BLOCO,
resp = MMG,
verbose = FALSE)
# Use o método print()
# ANOVA
print_tbl(reg_model$MMG$anova)
| SV | Df | Sum Sq | Mean Sq | F value | Pr(>F) |
|---|---|---|---|---|---|
| Total | 51 | 246217.905 | 4827.802 | NA | NA |
| GEN | 12 | 44633.375 | 3719.448 | 0.835 | 0.616 |
| ENV + (GEN x ENV) | 39 | 201584.529 | 5168.834 | NA | NA |
| ENV (linear) | 1 | 37012.734 | 37012.734 | NA | NA |
| GEN x ENV (linear) | 12 | 48764.695 | 4063.725 | 0.912 | 0.548 |
| Pooled deviation | 26 | 115807.101 | 4454.119 | NA | NA |
| H1 | 2 | 8958.150 | 4479.075 | 4.877 | 0.010 |
| H10 | 2 | 1618.745 | 809.372 | 0.881 | 0.418 |
| H11 | 2 | 1989.689 | 994.844 | 1.083 | 0.343 |
| H12 | 2 | 909.811 | 454.905 | 0.495 | 0.611 |
| H13 | 2 | 246.901 | 123.450 | 0.134 | 0.874 |
| H2 | 2 | 12810.743 | 6405.372 | 6.974 | 0.001 |
| H3 | 2 | 10966.021 | 5483.011 | 5.970 | 0.004 |
| H4 | 2 | 9729.718 | 4864.859 | 5.297 | 0.007 |
| H5 | 2 | 2213.292 | 1106.646 | 1.205 | 0.304 |
| H6 | 2 | 476.107 | 238.053 | 0.259 | 0.772 |
| H7 | 2 | 15616.320 | 7808.160 | 8.502 | 0.000 |
| H8 | 2 | 27920.883 | 13960.442 | 15.200 | 0.000 |
| H9 | 2 | 22350.721 | 11175.360 | 12.168 | 0.000 |
| Pooled error | 96 | 88170.644 | 918.444 | NA | NA |
# REGRESSÃO
print_tbl(reg_model$MMG$regression)
| GEN | b0 | b1 | t(b1=1) | pval_t | s2di | F(s2di=0) | pval_f | RMSE | R2 |
|---|---|---|---|---|---|---|---|---|---|
| H1 | 364.627 | -0.022 | -1.7987545 | 0.0751999942 | 1186.877 | 4.877 | 0.010 | 27.322 | 0.000 |
| H10 | 319.960 | 2.914 | 3.3700371 | 0.0010837020 | -36.357 | 0.881 | 0.418 | 11.614 | 0.937 |
| H11 | 332.915 | 1.456 | 0.8035295 | 0.4236538540 | 25.467 | 1.083 | 0.343 | 12.877 | 0.752 |
| H12 | 315.798 | 1.190 | 0.3344533 | 0.7387671092 | -154.513 | 0.495 | 0.611 | 8.707 | 0.816 |
| H13 | 340.238 | 1.186 | 0.3281713 | 0.7434968234 | -264.998 | 0.134 | 0.874 | 4.536 | 0.942 |
| H2 | 356.171 | -0.797 | -3.1642416 | 0.0020830059 | 1828.976 | 6.974 | 0.001 | 32.674 | 0.124 |
| H3 | 345.728 | -0.641 | -2.8896824 | 0.0047675367 | 1521.522 | 5.970 | 0.004 | 30.230 | 0.096 |
| H4 | 346.107 | 0.480 | -0.9147227 | 0.3626290955 | 1315.472 | 5.297 | 0.007 | 28.475 | 0.063 |
| H5 | 340.919 | 1.300 | 0.5286838 | 0.5982449757 | 62.734 | 1.205 | 0.304 | 13.581 | 0.685 |
| H6 | 363.105 | 1.993 | 1.7491926 | 0.0834537132 | -226.797 | 0.259 | 0.772 | 6.299 | 0.960 |
| H7 | 344.992 | 0.028 | -1.7107209 | 0.0903623671 | 2296.572 | 8.502 | 0.000 | 36.074 | 0.000 |
| H8 | 321.534 | 0.930 | -0.1226089 | 0.9026731286 | 4347.332 | 15.200 | 0.000 | 48.236 | 0.081 |
| H9 | 310.567 | 2.980 | 3.4866633 | 0.0007393608 | 3418.972 | 12.168 | 0.000 | 43.157 | 0.531 |
# Gráfico
p1 <- plot(reg_model)
p2 <- plot(reg_model,
x.lab = "Índice ambiental",
y.lab = "Massa de Mil Grãos (g)",
plot_theme = theme_metan_minimal())
p3 <- plot(reg_model, type = 2)
# reunir os plots
arrange_ggplot((p1 + p2), p3, ncol = 1,
guides = "collect",
tag_levels = "A",
tag_suffix = ")")
Índice de confiança genotípica
Annicchiarico (1992)4 propôs um método de estabilidade em que o parâmetro de estabilidade é medido pela superioridade do genótipo em relação à média de cada ambiente, de acordo com o seguinte modelo:
$$ {Z_ {ij}} = \frac {{{Y_ {ij}}}} {{{{\bar Y} _ {. J}}}} \times 100 $$ O índice de confiança genotípica do genótipo \(i\) (\(W_i\)) é então estimado da seguinte forma:
$$
W_i = Z_ {i.} / E - \alpha \times sd(Z_{i.})
$$
Onde \(\alpha\) é o quantil da distribuição normal padrão em uma dada probabilidade de erro (\(\alpha \approx 1,64 \alpha 0,05\)). O método é implementado usando a função Annicchiarico(). O índice de confiança é estimado considerando todos os ambientes, ambientes favoráveis (índice positivo) e ambientes desfavoráveis (índice negativo).
ann1 <- Annicchiarico(df_ge,
env = ENV,
gen = GEN,
rep = BLOCO,
resp = everything(),
verbose = FALSE)
# Wi
gmd(ann1) %>% print_tbl()
## Class of the model: Annicchiarico
## Variable extracted: Wi
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 99.317 | 100.724 | 98.724 | 101.093 | 98.724 | 96.970 | 103.686 | 99.249 | 100.459 | 91.829 |
| H10 | 88.284 | 84.746 | 97.320 | 94.301 | 91.868 | 97.185 | 88.240 | 92.785 | 86.375 | 95.169 |
| H11 | 91.871 | 86.621 | 97.448 | 95.913 | 94.219 | 98.024 | 95.253 | 90.163 | 94.645 | 95.045 |
| H12 | 90.999 | 81.972 | 91.209 | 96.745 | 93.802 | 90.189 | 84.650 | 98.175 | 90.942 | 90.930 |
| H13 | 94.449 | 91.370 | 95.457 | 99.175 | 95.254 | 95.548 | 93.585 | 102.707 | 99.215 | 94.549 |
| H2 | 98.144 | 89.381 | 98.173 | 98.973 | 93.203 | 96.692 | 98.078 | 95.687 | 95.690 | 97.699 |
| H3 | 96.619 | 91.333 | 90.269 | 98.041 | 94.393 | 94.104 | 89.909 | 92.443 | 93.396 | 89.245 |
| H4 | 97.694 | 95.229 | 100.839 | 97.376 | 95.165 | 100.665 | 100.550 | 91.344 | 95.472 | 101.952 |
| H5 | 99.462 | 95.434 | 100.237 | 98.267 | 96.982 | 102.230 | 102.157 | 93.330 | 97.287 | 100.812 |
| H6 | 97.707 | 99.749 | 97.930 | 102.562 | 99.271 | 98.663 | 96.757 | 97.304 | 103.558 | 89.813 |
| H7 | 92.264 | 91.922 | 96.634 | 98.231 | 98.731 | 96.497 | 90.193 | 97.969 | 93.005 | 95.396 |
| H8 | 87.109 | 78.137 | 93.663 | 95.019 | 92.440 | 94.651 | 80.447 | 95.255 | 83.628 | 90.798 |
| H9 | 90.185 | 87.555 | 94.189 | 92.809 | 94.146 | 95.306 | 76.645 | 91.159 | 79.349 | 88.262 |
# Ranques
gmd(ann1, "rank") %>% print_tbl()
## Class of the model: Annicchiarico
## Variable extracted: rank
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 2 | 1 | 3 | 2 | 3 | 6 | 1 | 2 | 2 | 8 |
| H10 | 12 | 11 | 7 | 12 | 13 | 5 | 10 | 9 | 11 | 5 |
| H11 | 9 | 10 | 6 | 10 | 8 | 4 | 6 | 13 | 7 | 6 |
| H12 | 10 | 12 | 12 | 9 | 10 | 13 | 11 | 3 | 10 | 9 |
| H13 | 7 | 6 | 9 | 3 | 5 | 9 | 7 | 1 | 3 | 7 |
| H2 | 3 | 8 | 4 | 4 | 11 | 7 | 4 | 6 | 5 | 3 |
| H3 | 6 | 7 | 13 | 7 | 7 | 12 | 9 | 10 | 8 | 12 |
| H4 | 5 | 4 | 1 | 8 | 6 | 2 | 3 | 11 | 6 | 1 |
| H5 | 1 | 3 | 2 | 5 | 4 | 1 | 2 | 8 | 4 | 2 |
| H6 | 4 | 2 | 5 | 1 | 1 | 3 | 5 | 5 | 1 | 11 |
| H7 | 8 | 5 | 8 | 6 | 2 | 8 | 8 | 4 | 9 | 4 |
| H8 | 13 | 13 | 11 | 11 | 12 | 11 | 12 | 7 | 12 | 10 |
| H9 | 11 | 9 | 10 | 13 | 9 | 10 | 13 | 12 | 13 | 13 |
# classificação dos ambientes
ann1$ALT_PLANT$environments %>% print_tbl()
| ENV | Y | index | class |
|---|---|---|---|
| A1 | 2.793 | 0.308 | favorable |
| A2 | 2.462 | -0.023 | unfavorable |
| A3 | 2.167 | -0.318 | unfavorable |
| A4 | 2.518 | 0.033 | favorable |
Índice de superioridade
A função superiority() implementa o método não paramétrico proposto por Lin e Binns (1988)5, que considera que uma medida de superioridade geral de cultivar para dados de ensaios multiambientes é definida como a soma do quadrado da distância média entre a resposta da cultivar e a resposta máxima calculada em todos os locais, de acordo com o seguinte modelo.
$$ P_i = \sum \limits_ {j = 1} ^ n {(y_ {ij} - y _ {. J}) ^ 2 / (2n)} $$ onde *n* é o número de ambientes
Semelhante ao índice de confiança genotípica, o índice de superioridade é calculado por todos os ambientes, favoráveis e desfavoráveis.
super <- superiority(df_ge,
env = ENV,
gen = GEN,
resp = everything(),
verbose = FALSE)
gmd(super) %>% print_tbl()
## Class of the model: superiority
## Variable extracted: Pi_a
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 0.026 | 0.007 | 1.287 | 1.418 | 3.055 | 1.533 | 332.263 | 1.987 | 873.628 | 6441.658 |
| H10 | 0.151 | 0.094 | 1.226 | 11.544 | 10.934 | 1.271 | 1147.318 | 3.693 | 3576.987 | 5152.155 |
| H11 | 0.116 | 0.085 | 1.296 | 9.327 | 9.207 | 1.153 | 918.019 | 4.384 | 2171.711 | 5686.694 |
| H12 | 0.103 | 0.106 | 3.130 | 8.863 | 9.456 | 3.591 | 1566.449 | 1.916 | 3447.948 | 6837.431 |
| H13 | 0.069 | 0.062 | 1.632 | 3.080 | 5.278 | 1.557 | 684.533 | 0.411 | 1798.489 | 4401.477 |
| H2 | 0.029 | 0.026 | 1.029 | 2.492 | 6.540 | 1.355 | 337.227 | 2.156 | 1365.866 | 4122.825 |
| H3 | 0.043 | 0.021 | 3.035 | 4.607 | 6.801 | 2.131 | 823.879 | 3.320 | 1745.780 | 8201.163 |
| H4 | 0.040 | 0.021 | 0.640 | 6.287 | 6.982 | 0.653 | 361.730 | 4.540 | 1487.961 | 3193.574 |
| H5 | 0.040 | 0.034 | 0.562 | 5.497 | 5.948 | 0.441 | 386.551 | 3.213 | 1824.117 | 2095.889 |
| H6 | 0.033 | 0.014 | 0.716 | 0.621 | 4.412 | 0.695 | 404.197 | 1.967 | 895.035 | 6042.938 |
| H7 | 0.109 | 0.064 | 1.178 | 6.168 | 4.808 | 1.050 | 1104.620 | 2.053 | 2627.212 | 6135.408 |
| H8 | 0.161 | 0.126 | 1.971 | 11.403 | 10.182 | 1.696 | 1788.539 | 3.018 | 4741.914 | 7287.795 |
| H9 | 0.124 | 0.068 | 2.031 | 15.070 | 8.573 | 1.667 | 2072.695 | 4.653 | 5702.604 | 7956.043 |
Estratificação ambiental
Um método que combina análise de estabilidade e estratificação ambiental usando análise fatorial foi proposto por Murakami e Cruz (2004)6. Este método é implementado com a função ge_factanal(), da seguinte forma:
fato <- ge_factanal(df_ge,
env = ENV,
gen = GEN,
rep = BLOCO,
resp = everything(),
verbose = FALSE,
mineval = 0.7)
# plot
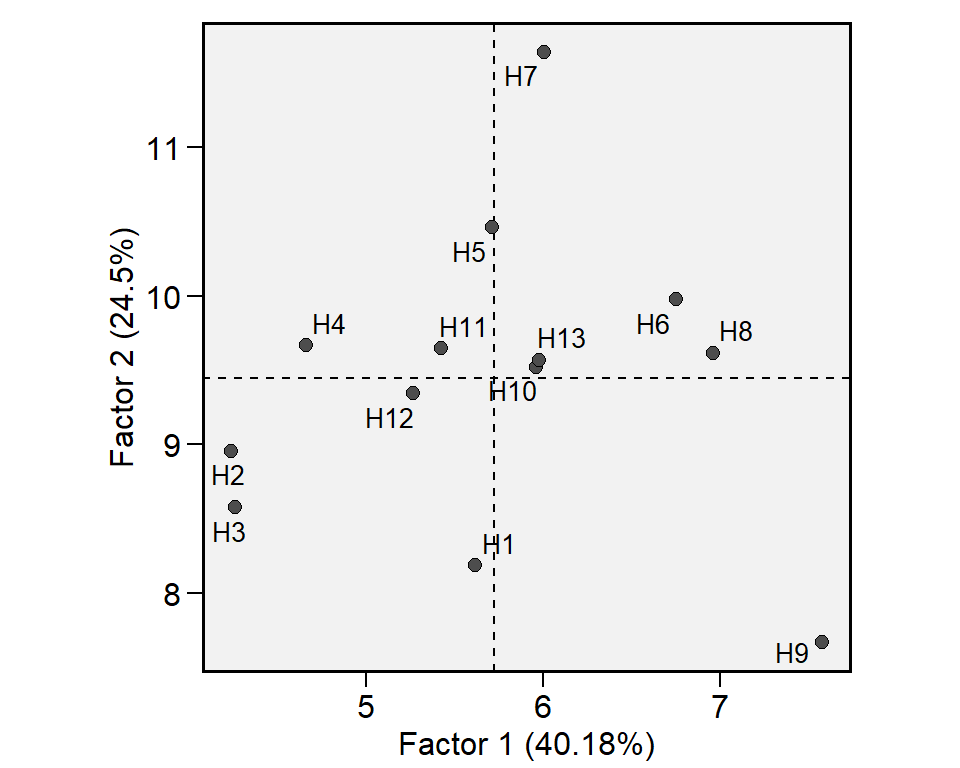
plot(fato, var = "MMG")
# Autovalores e variância
print_tbl(fato$MMG$PCA)
| PCA | Eigenvalues | Variance | Cumul_var |
|---|---|---|---|
| PC1 | 1.607 | 40.178 | 40.178 |
| PC2 | 0.980 | 24.500 | 64.678 |
| PC3 | 0.912 | 22.807 | 87.485 |
| PC4 | 0.501 | 12.515 | 100.000 |
# Cargas fatoriais após rotação varimax
print_tbl(fato$MMG$FA)
| Env | FA1 | FA2 | FA3 | Communality | Uniquenesses |
|---|---|---|---|---|---|
| A1 | 0.831 | -0.288 | -0.065 | 0.778 | 0.222 |
| A2 | -0.849 | -0.135 | -0.223 | 0.788 | 0.212 |
| A3 | -0.093 | 0.059 | -0.983 | 0.978 | 0.022 |
| A4 | -0.069 | 0.973 | -0.060 | 0.955 | 0.045 |
# Estratificação
print_tbl(fato$MMG$env_strat)
| Env | Factor | Mean | Min | Max | CV |
|---|---|---|---|---|---|
| A1 | FA1 | 360.337 | 325.013 | 413.088 | 8.096 |
| A2 | FA1 | 333.815 | 236.281 | 415.675 | 16.856 |
| A4 | FA2 | 342.796 | 293.694 | 391.762 | 7.228 |
| A3 | FA3 | 317.718 | 247.261 | 368.768 | 10.814 |
A função ge_stats()
A maneira mais fácil de calcular os índices de estabilidade mencionados acima é usando a função ge_stats(). É uma função “wrapper” que computa todos os índices de estabilidade de uma única vez. Para obter os resultados em um arquivo “pronto para ler”, use gmd().
stat_ge <- ge_stats(df_ge,
env = ENV,
gen = GEN,
rep = BLOCO,
resp = c(MMG, MGE))
## Evaluating trait MMG |====================== | 50% 00:00:03
Evaluating trait MGE |===========================================| 100% 00:00:05
# estatisticas
gmd(stat_ge, "stats") %>% print_tbl()
## Class of the model: ge_stats
## Variable extracted: stats
| var | GEN | Y | CV | ACV | POLAR | Var | Shukla | Wi_g | Wi_f | Wi_u | Ecoval | bij | Sij | R2 | ASV | SIPC | EV | ZA | WAAS | WAASB | HMGV | RPGV | HMRPGV | Pi_a | Pi_f | Pi_u | Gai | S1 | S2 | S3 | S6 | N1 | N2 | N3 | N4 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| MGE | H1 | 183.747 | 10.498 | 12.083 | -0.247 | 1116.387 | 11.555 | 99.657 | 98.888 | 103.555 | 414.699 | 0.844 | -77.149 | 0.907 | 2.707 | 1.573 | 0.008 | 0.071 | 0.782 | 1.155 | 179.691 | 1.051 | 1.050 | 332.263 | 275.953 | 388.573 | 182.951 | 0.167 | 3.583 | 0.081 | 0.162 | 1.25 | 0.250 | 0.345 | 0.035 |
| MGE | H10 | 163.893 | 19.491 | 17.377 | 0.069 | 3061.262 | 160.408 | 79.699 | 92.027 | 73.624 | 1548.269 | 1.395 | 18.268 | 0.904 | 2.809 | 2.830 | 0.050 | 0.091 | 0.895 | 1.160 | 161.696 | 0.955 | 0.951 | 1147.318 | 493.895 | 1800.741 | 161.345 | 1.333 | 11.667 | 5.556 | 1.778 | 2.50 | 0.263 | 0.311 | 0.140 |
| MGE | H11 | 167.240 | 12.166 | 11.347 | -0.301 | 1241.912 | -20.805 | 93.105 | 90.933 | 94.996 | 168.262 | 0.917 | -105.803 | 0.963 | 0.994 | 0.981 | 0.006 | 0.032 | 0.321 | 0.421 | 166.502 | 0.974 | 0.974 | 918.019 | 655.031 | 1181.006 | 166.278 | 0.333 | 0.917 | 1.667 | 0.952 | 0.75 | 0.088 | 0.095 | 0.038 |
| MGE | H12 | 157.474 | 13.961 | 11.385 | -0.298 | 1450.104 | 287.701 | 75.021 | 86.923 | 62.608 | 2517.655 | 0.715 | 232.821 | 0.501 | 8.213 | 3.614 | 0.040 | 0.187 | 2.150 | 2.122 | 158.882 | 0.932 | 0.928 | 1566.449 | 939.068 | 2193.831 | 156.298 | 0.500 | 11.333 | 6.294 | 2.118 | 2.50 | 0.250 | 0.299 | 0.051 |
| MGE | H13 | 179.838 | 16.076 | 17.635 | 0.082 | 2507.555 | 790.862 | 77.725 | 68.274 | 84.912 | 6349.416 | 0.637 | 835.854 | 0.230 | 6.391 | 6.619 | 0.296 | 0.202 | 1.952 | 1.736 | 176.064 | 1.036 | 1.025 | 684.533 | 749.402 | 619.664 | 178.152 | 2.000 | 30.333 | 9.059 | 1.882 | 4.50 | 1.000 | 0.867 | 0.364 |
| MGE | H2 | 186.999 | 15.420 | 18.457 | 0.121 | 2494.460 | 783.810 | 82.899 | 87.203 | 95.440 | 6295.715 | 0.639 | 827.773 | 0.233 | 14.353 | 4.516 | 0.088 | 0.282 | 3.366 | 2.858 | 181.481 | 1.067 | 1.059 | 337.227 | 561.094 | 113.359 | 185.339 | 0.833 | 24.917 | 6.135 | 1.351 | 3.75 | 1.071 | 0.910 | 0.175 |
| MGE | H3 | 169.456 | 17.398 | 16.711 | 0.035 | 2607.508 | 457.681 | 78.288 | 74.688 | 82.277 | 3812.115 | 0.970 | 505.775 | 0.513 | 11.355 | 4.243 | 0.061 | 0.240 | 2.813 | 1.892 | 167.189 | 0.984 | 0.979 | 823.879 | 998.956 | 648.801 | 167.511 | 0.000 | 12.667 | 8.182 | 2.182 | 3.00 | 0.353 | 0.363 | 0.000 |
| MGE | H4 | 184.260 | 14.858 | 17.208 | 0.061 | 2248.709 | 210.678 | 92.036 | 95.538 | 84.770 | 1931.096 | 1.064 | 189.975 | 0.716 | 5.337 | 4.136 | 0.075 | 0.160 | 1.694 | 1.633 | 178.833 | 1.051 | 1.048 | 361.730 | 245.252 | 478.208 | 182.562 | 0.667 | 10.000 | 1.378 | 0.757 | 2.50 | 0.556 | 0.577 | 0.140 |
| MGE | H5 | 183.635 | 8.399 | 9.653 | -0.442 | 713.629 | 119.666 | 95.631 | 87.860 | 109.197 | 1238.005 | 0.606 | -33.097 | 0.731 | 1.727 | 1.796 | 0.022 | 0.054 | 0.519 | 1.184 | 180.090 | 1.052 | 1.050 | 386.551 | 402.287 | 370.815 | 183.123 | 0.167 | 13.667 | 3.757 | 1.189 | 2.00 | 0.500 | 0.674 | 0.035 |
| MGE | H6 | 187.999 | 23.075 | 27.950 | 0.482 | 5645.769 | 938.289 | 80.515 | 80.748 | 70.620 | 7472.134 | 1.609 | 852.492 | 0.652 | 13.863 | 4.217 | 0.082 | 0.268 | 3.218 | 3.098 | 179.051 | 1.063 | 1.053 | 404.197 | 308.903 | 499.492 | 184.086 | 0.333 | 34.000 | 10.250 | 2.250 | 5.00 | 0.833 | 0.842 | 0.056 |
| MGE | H7 | 171.051 | 15.553 | 15.256 | -0.044 | 2123.393 | 616.384 | 77.098 | 75.298 | 71.682 | 5020.702 | 0.658 | 624.746 | 0.290 | 11.555 | 5.093 | 0.079 | 0.263 | 3.027 | 2.351 | 169.147 | 0.994 | 0.987 | 1104.620 | 632.640 | 1576.600 | 169.516 | 0.333 | 30.917 | 10.571 | 2.286 | 4.75 | 0.633 | 0.688 | 0.048 |
| MGE | H8 | 159.860 | 24.757 | 20.877 | 0.228 | 4698.738 | 971.105 | 63.670 | 92.782 | 47.656 | 7722.038 | 1.247 | 1114.690 | 0.471 | 16.164 | 5.511 | 0.115 | 0.328 | 3.887 | 2.995 | 157.575 | 0.938 | 0.924 | 1788.539 | 370.246 | 3206.831 | 155.981 | 0.333 | 24.250 | 5.333 | 1.333 | 2.75 | 0.344 | 0.533 | 0.042 |
| MGE | H9 | 152.753 | 28.202 | 21.486 | 0.253 | 5567.447 | 807.372 | 61.157 | 93.700 | 55.597 | 6475.147 | 1.699 | 603.283 | 0.737 | 12.230 | 4.923 | 0.077 | 0.267 | 3.106 | 3.324 | 151.076 | 0.902 | 0.890 | 2072.695 | 434.254 | 3711.136 | 148.265 | 1.000 | 26.250 | 12.400 | 2.800 | 4.25 | 0.425 | 0.493 | 0.111 |
| MMG | H1 | 364.627 | 8.653 | 11.222 | 0.026 | 2986.494 | 1428.010 | 89.756 | 84.496 | 115.774 | 11929.793 | -0.022 | 1186.877 | 0.000 | 7.758 | 9.280 | 0.115 | 0.233 | 2.723 | 3.488 | 357.771 | 1.062 | 1.057 | 873.628 | 1568.453 | 178.803 | 363.550 | 1.667 | 20.250 | 7.865 | 1.568 | 3.25 | 1.083 | 0.820 | 0.351 |
| MMG | H10 | 319.960 | 16.732 | 13.805 | 0.206 | 8598.665 | 1443.749 | 75.316 | 99.000 | 66.671 | 12049.653 | 2.914 | -36.357 | 0.937 | 6.579 | 8.775 | 0.119 | 0.196 | 2.154 | 2.473 | 318.632 | 0.953 | 0.946 | 3576.987 | 976.033 | 6177.942 | 316.356 | 1.667 | 24.917 | 7.435 | 1.913 | 3.75 | 0.469 | 0.524 | 0.202 |
| MMG | H11 | 332.915 | 8.971 | 8.492 | -0.216 | 2676.187 | 200.613 | 89.534 | 95.619 | 83.154 | 2582.691 | 1.456 | 25.467 | 0.752 | 3.549 | 4.588 | 0.024 | 0.110 | 1.260 | 0.801 | 332.381 | 0.986 | 0.984 | 2171.711 | 1379.858 | 2963.563 | 331.848 | 1.167 | 11.333 | 3.231 | 1.231 | 2.00 | 0.286 | 0.389 | 0.156 |
| MMG | H12 | 315.798 | 7.420 | 5.850 | -0.540 | 1647.118 | -5.568 | 87.709 | 89.102 | 86.260 | 1012.547 | 1.190 | -154.513 | 0.816 | 0.981 | 2.640 | 0.012 | 0.053 | 0.558 | 1.589 | 319.446 | 0.946 | 0.946 | 3447.948 | 2456.959 | 4438.937 | 315.125 | 0.667 | 3.667 | 0.846 | 0.923 | 1.50 | 0.143 | 0.154 | 0.062 |
| MMG | H13 | 340.238 | 6.390 | 6.522 | -0.445 | 1418.101 | -93.119 | 97.470 | 100.300 | 96.135 | 345.814 | 1.186 | -264.998 | 0.942 | 2.451 | 0.870 | 0.001 | 0.038 | 0.514 | 0.423 | 338.968 | 1.003 | 1.003 | 1798.489 | 1021.110 | 2575.868 | 339.724 | 0.000 | 2.667 | 0.667 | 0.533 | 1.00 | 0.154 | 0.218 | 0.000 |
| MMG | H2 | 356.171 | 11.316 | 13.531 | 0.188 | 4873.365 | 2751.227 | 81.518 | 87.132 | 98.505 | 22006.597 | -0.797 | 1828.976 | 0.124 | 19.026 | 9.104 | 0.081 | 0.338 | 4.407 | 3.857 | 350.636 | 1.044 | 1.035 | 1365.866 | 2601.310 | 130.422 | 354.565 | 1.000 | 30.333 | 9.733 | 2.133 | 4.50 | 0.692 | 0.734 | 0.154 |
| MMG | H3 | 345.728 | 10.622 | 11.458 | 0.044 | 4045.579 | 2308.528 | 80.412 | 87.758 | 96.442 | 18635.273 | -0.641 | 1521.522 | 0.096 | 17.311 | 8.814 | 0.073 | 0.316 | 4.097 | 3.080 | 342.651 | 1.019 | 1.011 | 1745.780 | 3162.932 | 328.628 | 344.351 | 1.333 | 27.667 | 12.600 | 2.720 | 4.50 | 0.562 | 0.588 | 0.172 |
| MMG | H4 | 346.107 | 9.816 | 10.628 | -0.021 | 3462.326 | 1240.023 | 85.656 | 87.827 | 83.354 | 10498.197 | 0.480 | 1315.472 | 0.063 | 12.974 | 6.624 | 0.041 | 0.237 | 3.070 | 2.769 | 342.804 | 1.018 | 1.014 | 1487.961 | 1831.223 | 1144.699 | 344.861 | 0.500 | 14.250 | 3.600 | 1.333 | 2.75 | 0.458 | 0.503 | 0.077 |
| MMG | H5 | 340.919 | 8.196 | 8.423 | -0.223 | 2342.327 | 185.815 | 92.522 | 93.187 | 93.797 | 2470.003 | 1.300 | 62.734 | 0.685 | 0.984 | 3.799 | 0.030 | 0.072 | 0.738 | 0.476 | 338.999 | 1.005 | 1.004 | 1824.117 | 932.079 | 2716.156 | 340.044 | 1.333 | 12.917 | 3.452 | 1.097 | 2.25 | 0.321 | 0.498 | 0.213 |
| MMG | H6 | 363.105 | 9.968 | 12.742 | 0.136 | 3930.178 | 292.999 | 98.565 | 101.865 | 102.976 | 3286.247 | 1.993 | -226.797 | 0.960 | 5.731 | 4.614 | 0.022 | 0.134 | 1.636 | 0.591 | 355.852 | 1.055 | 1.054 | 895.035 | 162.728 | 1627.343 | 361.789 | 0.000 | 10.250 | 1.000 | 0.600 | 1.75 | 0.438 | 0.693 | 0.000 |
| MMG | H7 | 344.992 | 12.075 | 12.930 | 0.149 | 5206.204 | 2265.054 | 79.957 | 80.262 | 70.786 | 18304.208 | 0.028 | 2296.572 | 0.000 | 9.611 | 12.375 | 0.172 | 0.300 | 3.442 | 2.550 | 341.194 | 1.016 | 1.008 | 2627.212 | 1406.081 | 3848.343 | 343.060 | 0.167 | 27.333 | 9.061 | 2.061 | 4.50 | 0.818 | 0.787 | 0.029 |
| MMG | H8 | 321.534 | 18.071 | 15.165 | 0.288 | 10128.429 | 3529.663 | 67.320 | 95.892 | 45.138 | 27934.690 | 0.930 | 4347.332 | 0.081 | 19.829 | 11.185 | 0.134 | 0.384 | 4.909 | 4.496 | 319.330 | 0.960 | 0.945 | 4741.914 | 1327.984 | 8155.844 | 317.081 | 0.833 | 26.000 | 8.154 | 1.846 | 3.50 | 0.500 | 0.589 | 0.111 |
| MMG | H9 | 310.567 | 23.426 | 17.434 | 0.409 | 15879.870 | 4262.573 | 62.157 | 66.471 | 58.698 | 33516.082 | 2.980 | 3418.972 | 0.531 | 21.320 | 13.615 | 0.175 | 0.436 | 5.499 | 5.127 | 308.772 | 0.931 | 0.916 | 5702.604 | 2404.298 | 9000.910 | 304.625 | 0.667 | 23.583 | 18.000 | 3.200 | 3.75 | 0.341 | 0.467 | 0.074 |
# Ranques
gmd(stat_ge, "ranks") %>% print_tbl()
## Class of the model: ge_stats
## Variable extracted: ranks
| var | GEN | Y_R | CV_R | ACV_R | POLAR_R | Var_R | Shukla_R | Wi_g_R | Wi_f_R | Wi_u_R | Ecoval_R | Sij_R | R2_R | ASV_R | SIPC_R | EV_R | ZA_R | WAAS_R | WAASB_R | HMGV_R | RPGV_R | HMRPGV_R | Pi_a_R | Pi_f_R | Pi_u_R | Gai_R | S1_R | S2_R | S3_R | S6_R | N1_R | N2_R | N3_R | N4_R |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| MGE | H1 | 4 | 2 | 4 | 4 | 2 | 2 | 1 | 1 | 2 | 2 | 3 | 2 | 3 | 2 | 2 | 3 | 3 | 2 | 3 | 5 | 4 | 1 | 2 | 3 | 4 | 2.5 | 2 | 1 | 1 | 2.0 | 2.5 | 4 | 2.5 |
| MGE | H10 | 10 | 10 | 8 | 8 | 10 | 4 | 7 | 5 | 8 | 4 | 1 | 3 | 4 | 4 | 5 | 4 | 4 | 3 | 10 | 10 | 10 | 10 | 7 | 10 | 10 | 12.0 | 5 | 6 | 7 | 5.0 | 4.0 | 3 | 10.5 |
| MGE | H11 | 9 | 3 | 2 | 2 | 3 | 1 | 3 | 6 | 4 | 1 | 4 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 9 | 9 | 9 | 8 | 10 | 8 | 9 | 5.5 | 1 | 3 | 3 | 1.0 | 1.0 | 1 | 4.0 |
| MGE | H12 | 12 | 4 | 3 | 3 | 4 | 6 | 11 | 9 | 11 | 6 | 6 | 9 | 7 | 5 | 4 | 6 | 7 | 8 | 11 | 12 | 11 | 11 | 12 | 11 | 11 | 8.0 | 4 | 8 | 9 | 5.0 | 2.5 | 2 | 7.0 |
| MGE | H13 | 6 | 8 | 9 | 9 | 8 | 10 | 9 | 13 | 5 | 10 | 11 | 13 | 6 | 13 | 13 | 7 | 6 | 6 | 6 | 6 | 6 | 6 | 11 | 6 | 6 | 13.0 | 11 | 10 | 8 | 11.0 | 12.0 | 12 | 13.0 |
| MGE | H2 | 2 | 6 | 10 | 10 | 7 | 9 | 5 | 8 | 3 | 9 | 10 | 12 | 12 | 9 | 11 | 12 | 12 | 10 | 1 | 1 | 1 | 2 | 8 | 1 | 1 | 10.0 | 9 | 7 | 6 | 9.0 | 13.0 | 13 | 12.0 |
| MGE | H3 | 8 | 9 | 6 | 6 | 9 | 7 | 8 | 12 | 7 | 7 | 7 | 8 | 8 | 8 | 6 | 8 | 8 | 7 | 8 | 8 | 8 | 7 | 13 | 7 | 8 | 1.0 | 6 | 9 | 10 | 8.0 | 6.0 | 5 | 1.0 |
| MGE | H4 | 3 | 5 | 7 | 7 | 6 | 5 | 4 | 2 | 6 | 5 | 5 | 6 | 5 | 6 | 7 | 5 | 5 | 5 | 5 | 4 | 5 | 3 | 1 | 4 | 5 | 9.0 | 3 | 2 | 2 | 5.0 | 9.0 | 8 | 10.5 |
| MGE | H5 | 5 | 1 | 1 | 1 | 1 | 3 | 2 | 7 | 1 | 3 | 2 | 5 | 2 | 3 | 3 | 2 | 2 | 4 | 2 | 3 | 3 | 4 | 5 | 2 | 3 | 2.5 | 7 | 4 | 4 | 3.0 | 8.0 | 9 | 2.5 |
| MGE | H6 | 1 | 11 | 13 | 13 | 13 | 12 | 6 | 10 | 10 | 12 | 12 | 7 | 11 | 7 | 10 | 11 | 11 | 12 | 4 | 2 | 2 | 5 | 3 | 5 | 2 | 5.5 | 13 | 11 | 11 | 13.0 | 11.0 | 11 | 8.0 |
| MGE | H7 | 7 | 7 | 5 | 5 | 5 | 8 | 10 | 11 | 9 | 8 | 9 | 11 | 9 | 11 | 9 | 9 | 9 | 9 | 7 | 7 | 7 | 9 | 9 | 9 | 7 | 5.5 | 12 | 12 | 12 | 12.0 | 10.0 | 10 | 6.0 |
| MGE | H8 | 11 | 12 | 11 | 11 | 11 | 13 | 12 | 4 | 13 | 13 | 13 | 10 | 13 | 12 | 12 | 13 | 13 | 11 | 12 | 11 | 12 | 12 | 4 | 12 | 12 | 5.5 | 8 | 5 | 5 | 7.0 | 5.0 | 7 | 5.0 |
| MGE | H9 | 13 | 13 | 12 | 12 | 12 | 11 | 13 | 3 | 12 | 11 | 8 | 4 | 10 | 10 | 8 | 10 | 10 | 13 | 13 | 13 | 13 | 13 | 6 | 13 | 13 | 11.0 | 10 | 13 | 13 | 10.0 | 7.0 | 6 | 9.0 |
| MMG | H1 | 1 | 4 | 6 | 6 | 5 | 7 | 4 | 11 | 1 | 7 | 7 | 12 | 7 | 10 | 9 | 7 | 7 | 10 | 1 | 1 | 1 | 1 | 8 | 2 | 1 | 12.5 | 7 | 8 | 7 | 7.0 | 13.0 | 13 | 13.0 |
| MMG | H10 | 11 | 11 | 11 | 11 | 11 | 8 | 11 | 3 | 11 | 8 | 2 | 3 | 6 | 7 | 10 | 6 | 6 | 6 | 12 | 11 | 10 | 11 | 3 | 11 | 11 | 12.5 | 9 | 7 | 9 | 9.5 | 8.0 | 7 | 11.0 |
| MMG | H11 | 9 | 5 | 4 | 4 | 4 | 4 | 5 | 5 | 9 | 4 | 1 | 5 | 4 | 4 | 4 | 4 | 4 | 4 | 9 | 9 | 9 | 8 | 6 | 8 | 9 | 9.0 | 4 | 4 | 5 | 4.0 | 3.0 | 3 | 9.0 |
| MMG | H12 | 12 | 2 | 1 | 1 | 2 | 2 | 6 | 7 | 7 | 2 | 4 | 4 | 1 | 2 | 2 | 2 | 2 | 5 | 10 | 12 | 11 | 10 | 11 | 10 | 12 | 5.5 | 2 | 2 | 3 | 2.0 | 1.0 | 1 | 4.0 |
| MMG | H13 | 8 | 1 | 2 | 2 | 1 | 1 | 2 | 2 | 5 | 1 | 6 | 2 | 3 | 1 | 1 | 1 | 1 | 1 | 8 | 8 | 8 | 6 | 4 | 6 | 8 | 1.5 | 1 | 1 | 1 | 1.0 | 2.0 | 2 | 1.5 |
| MMG | H2 | 3 | 9 | 10 | 10 | 9 | 11 | 8 | 10 | 3 | 11 | 10 | 8 | 11 | 9 | 8 | 11 | 11 | 11 | 3 | 3 | 3 | 3 | 12 | 1 | 3 | 8.0 | 13 | 11 | 11 | 12.0 | 11.0 | 11 | 8.0 |
| MMG | H3 | 5 | 8 | 7 | 7 | 8 | 10 | 9 | 9 | 4 | 10 | 9 | 9 | 10 | 8 | 7 | 10 | 10 | 9 | 5 | 4 | 5 | 5 | 13 | 3 | 5 | 10.5 | 12 | 12 | 12 | 12.0 | 10.0 | 8 | 10.0 |
| MMG | H4 | 4 | 6 | 5 | 5 | 6 | 6 | 7 | 8 | 8 | 6 | 8 | 11 | 9 | 6 | 6 | 8 | 8 | 8 | 4 | 5 | 4 | 4 | 9 | 4 | 4 | 4.0 | 6 | 6 | 6 | 6.0 | 7.0 | 6 | 6.0 |
| MMG | H5 | 7 | 3 | 3 | 3 | 3 | 3 | 3 | 6 | 6 | 3 | 3 | 6 | 2 | 3 | 5 | 3 | 3 | 2 | 7 | 7 | 7 | 7 | 2 | 7 | 7 | 10.5 | 5 | 5 | 4 | 5.0 | 4.0 | 5 | 12.0 |
| MMG | H6 | 2 | 7 | 8 | 8 | 7 | 5 | 1 | 1 | 2 | 5 | 5 | 1 | 5 | 5 | 3 | 5 | 5 | 3 | 2 | 2 | 2 | 2 | 1 | 5 | 2 | 1.5 | 3 | 3 | 2 | 3.0 | 6.0 | 10 | 1.5 |
| MMG | H7 | 6 | 10 | 9 | 9 | 10 | 9 | 10 | 12 | 10 | 9 | 11 | 13 | 8 | 12 | 12 | 9 | 9 | 7 | 6 | 6 | 6 | 9 | 7 | 9 | 6 | 3.0 | 11 | 10 | 10 | 12.0 | 12.0 | 12 | 3.0 |
| MMG | H8 | 10 | 12 | 12 | 12 | 12 | 12 | 12 | 4 | 13 | 12 | 13 | 10 | 12 | 11 | 11 | 12 | 12 | 12 | 11 | 10 | 12 | 12 | 5 | 12 | 10 | 7.0 | 10 | 9 | 8 | 8.0 | 9.0 | 9 | 7.0 |
| MMG | H9 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 12 | 13 | 12 | 7 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 13 | 10 | 13 | 13 | 5.5 | 8 | 13 | 13 | 9.5 | 5.0 | 4 | 5.0 |
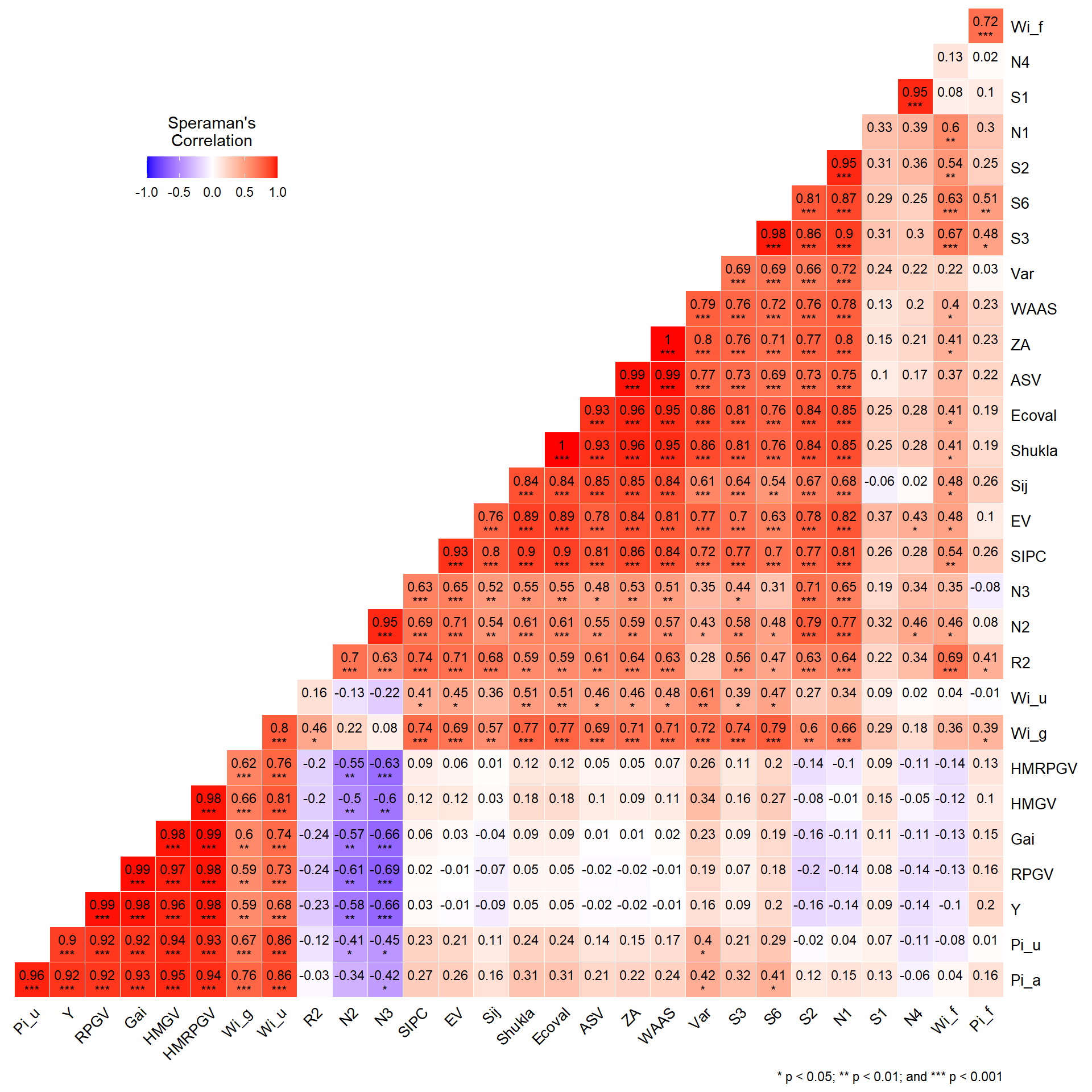
Também é possível obter a correlação de postos de Spearman entre os índices de estabilidade usando corr_stab_ind().
corr_stab_ind(stat_ge)
-
Robertson, A. (1959). Experimental design on the measurement of heritabilities and genetic correlations: Biometrical genetics. In Biometrical genetics. Pergamon. ↩︎
-
CRUZ, C.D.; CASTOLDI, F.L. Decomposição da interação genótipos x ambientes em partes simples e complexa. Revista Ceres, v.38, n.219, p.422-430, 1991. ↩︎
-
Eberhart, S. A., & Russell, W. A. (1966). Stability parameters for comparing varieties. Crop Science, 6(1), 36–40. https://doi.org/10.2135/cropsci1966.0011183X000600010011x ↩︎
-
Annicchiarico, P. (1992). Cultivar adaptation and recommendation from alfalfa trials in Northern Italy. Journal of Genetics and Breeding, 46, 269–278. ↩︎
-
Lin, C.S.; & Binns, M.R. (1988). A superiority measure of cultivar performance for cultivar x location data. Canadian Journal of Plant Science, 68, 193-198. https://cdnsciencepub.com/doi/abs/10.4141/cjps88-018 ↩︎
-
Murakami, D. M., & Cruz, C. D. (2004). Proposal of methodologies for environment stratification and analysis of genotype adaptability. Cropp Breeding and Applied Biotechnology, 4(1), 7–11. https://doi.org/10.12702/1984-7033.v04n01a02 ↩︎