Combinando AMMI e BLUP
library(metan)
## Registered S3 method overwritten by 'GGally':
## method from
## +.gg ggplot2
## |=========================================================|
## | Multi-Environment Trial Analysis (metan) v1.15.0 |
## | Author: Tiago Olivoto |
## | Type 'citation('metan')' to know how to cite metan |
## | Type 'vignette('metan_start')' for a short tutorial |
## | Visit 'https://bit.ly/pkgmetan' for a complete tutorial |
## |=========================================================|
library(rio)
library(ggforce)
## Carregando pacotes exigidos: ggplot2
# gerar tabelas html
print_tbl <- function(table, digits = 3, ...){
knitr::kable(table, booktabs = TRUE, digits = digits, ...)
}
df_ge <- import("http://bit.ly/df_ge", setclass = "tbl")
Índices univariados
A função waasb() calcula o WAASB, acrônimo para Weighted Average of the Absolute Scores obtidos pela decomposição por valores singulares dos BLUPs para interação genótipo-ambiente obtidos por um Modelo Linear de Efeito Misto1. O índice WAASB para um determinado genótipo ((i\)) é dado por:
$$ WAASB_i = \sum_ {k = 1} ^ {p} | IPCA_ {ik} \times EP_k | / \sum_ {k = 1} ^ {p} EP_k $$
onde \(WAASB_i\) é a média ponderada dos scores absolutos do \(i\)-ésimo genótipo; \(IPCA_{ik}\) são os scores do \(i\)-ésimo genótipo no \(k\)-ésimo IPCA; \(EP_k\) é a variância explicada no \(k\)-ésimo IPCA para \(k = 1,2, .., p\), \(p = min (g-1; e-1)\). O modelo é ajustado com a função waasb(). Tanto waasb() quanto gamem_met() ajustam o mesmo modelo.
model_waasb <-
waasb(df_ge,
env = ENV,
gen = GEN,
rep = BLOCO,
resp = everything(),
verbose = FALSE)
# índice WAASB
waasb_ind <- gmd(model_waasb, "WAASB")
## Class of the model: waasb
## Variable extracted: WAASB
print_tbl(waasb_ind)
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 0.319 | 0.349 | 0.084 | 0.695 | 0.802 | 0.156 | 1.155 | 0.249 | 3.488 | 2.548 |
| H10 | 0.287 | 0.232 | 0.163 | 0.879 | 0.871 | 0.184 | 1.160 | 0.392 | 2.473 | 0.701 |
| H11 | 0.210 | 0.210 | 0.122 | 0.579 | 0.487 | 0.160 | 0.421 | 0.597 | 0.801 | 0.777 |
| H12 | 0.298 | 0.334 | 0.490 | 0.344 | 0.574 | 0.553 | 2.122 | 0.165 | 1.589 | 1.893 |
| H13 | 0.258 | 0.200 | 0.308 | 0.758 | 0.769 | 0.350 | 1.736 | 0.730 | 0.423 | 4.467 |
| H2 | 0.312 | 0.332 | 0.210 | 0.990 | 0.972 | 0.377 | 2.858 | 0.634 | 3.857 | 2.249 |
| H3 | 0.340 | 0.322 | 0.527 | 0.364 | 0.541 | 0.386 | 1.892 | 0.466 | 3.080 | 2.065 |
| H4 | 0.268 | 0.252 | 0.326 | 0.321 | 0.506 | 0.329 | 1.633 | 0.443 | 2.769 | 0.095 |
| H5 | 0.171 | 0.101 | 0.301 | 0.465 | 0.365 | 0.310 | 1.184 | 0.573 | 0.476 | 2.148 |
| H6 | 0.232 | 0.170 | 0.601 | 0.528 | 0.688 | 0.536 | 3.098 | 0.317 | 0.591 | 3.901 |
| H7 | 0.209 | 0.154 | 0.442 | 0.301 | 0.408 | 0.469 | 2.351 | 0.226 | 2.550 | 1.016 |
| H8 | 0.334 | 0.336 | 0.456 | 0.640 | 0.917 | 0.466 | 2.995 | 0.273 | 4.496 | 2.508 |
| H9 | 0.208 | 0.071 | 0.374 | 0.913 | 0.644 | 0.330 | 3.324 | 0.415 | 5.127 | 3.357 |
Biplots
Como o índice WAASB é baseado em decomposição por valores singulares, é possível obter os mesmos biplots utilizados na análise AMMI convencional
p1 <- plot_scores(model_waasb, var = 9)
p2 <- plot_scores(model_waasb, type = 2, var = 9)
p1 + p2
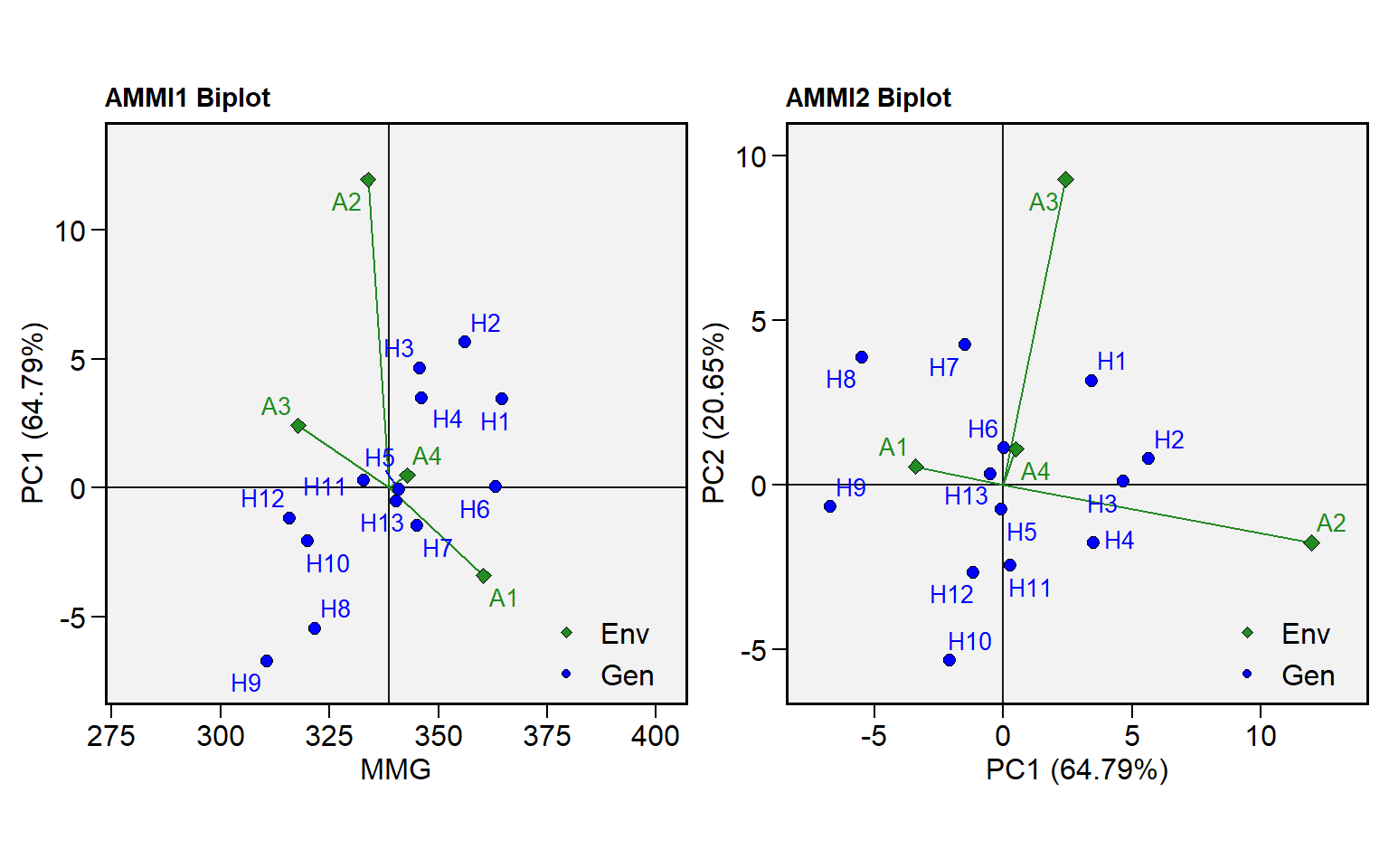
No método WAASB, o seguinte biplot representa quatro classificações relativas à interpretação conjunta de desempenho médio e estabilidade (para genótipos) ou discriminação (ambientes).
-
Quadrante I. Os genótipos incluídos neste quadrante podem ser considerados genótipos instáveis e com produtividade abaixo da média geral. Ambientes neste quadrante apresentam alta capacidade de discriminação.
-
Quadrante II. Neste quadrante estão incluídos os genótipos instáveis, embora com produtividade acima da média geral. Os ambientes incluídos neste quadrante merecem atenção especial, pois, além de proporcionarem altas magnitudes da variável resposta, apresentam boa capacidade de discriminação.
-
Quadrante III. Os genótipos deste quadrante apresentam baixa produtividade, mas podem ser considerados estáveis, devido aos menores valores de WAASB. Quanto menor esse valor, mais estável o genótipo pode ser considerado. Os ambientes incluídos neste quadrante podem ser considerados pouco produtivos e com baixa capacidade de discriminação.
-
Quadrante IV. Os genótipos dentro deste quadrante são altamente produtivos e amplamente adaptados devido à alta magnitude da variável de resposta e baixos valores do índice WAASB.
p3 <- plot_scores(model_waasb, type = 3, var = 9)
p4 <- plot_scores(model_waasb,
type = 3,
var = 9,
highlight = c("H1", "H6"),
plot_theme = theme_metan_minimal(),
title = FALSE)
arrange_ggplot(p3, p4, tag_levels = "a", guides = "collect")
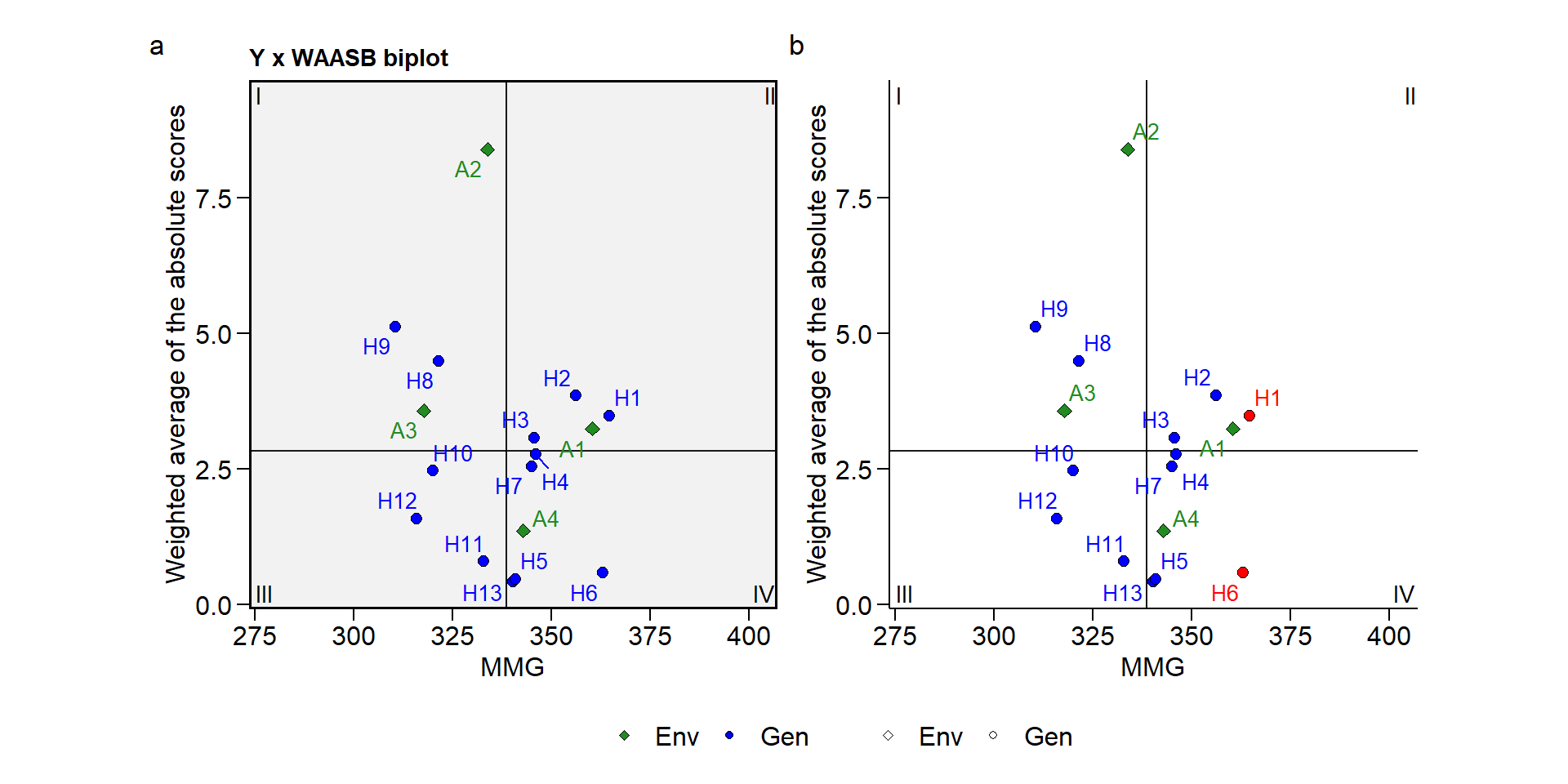
# extendendo o plot
desc <- c("Esses híbridos têm rendimento de grãos acima da média geral. \ N
Eles são mais estáveis do que aqueles acima da linha horizontal")
plot_scores(model_waasb,
type = 3,
var = 9,
x.lab = "Massa de mil grãos (g)",
y.lab = "Média poderada dos escores absolutos (WAASB)",
col.segm.env = "transparent") +
geom_mark_rect(aes(filter = Code %in% c("H13", "H4", "H6"),
label = "Descrição",
description = desc),
label.fontsize = 9,
show.legend = F,
con.cap = 0,
con.colour = "red",
color = "red",
expand = 0.015,
label.buffer = unit(2, "cm"))+
theme_gray()+
theme(legend.position = c(0.1, 0.9),
legend.background = element_blank(),
legend.title = element_blank(),
aspect.ratio = 1)

A seleção simultânea para desempenho médio e estabilidade é baseada no índice WAASBY1. Este índice considera a estabilidade (WAASB) e o desempenho médio (variável dependente, Y) para classificação de genótipos considerando o seguinte modelo:
$$ WAASB{Y_i} = \frac {{\left ({r {G_i} \times {\theta _Y}} \right) + \left ({r {W_i} \times {\theta _S}} \right)}} {{{\theta _Y} + {\theta _S}}} $$
onde \(WAASBY_i\) é o índice de seleção simultânea para o \(i\)-ésimo genótipo que pondera entre desempenho e estabilidade; \(rY_i\) e \(rW_i\) são os valores reescalados (0-100) para a variável dependente e WAASB, respectivamente; \(\theta _Y\) e \(\theta_S\) são os pesos da variável dependente e WAASB, respectivamente. Os valores redimensionados são usados para tornar WAASB e Y diretamente comparáveis. Os valores máximo e mínimo para redimensionar a variável dependente dependerão do objetivo da seleção. Por exemplo, assumindo que o valor mais alto para a variável dependente é melhor, digamos, para rendimento de grãos, o genótipo com a média mais alta terá \(rY_i = 100\) após o reescalonamento. Por outro lado, se o valor mais baixo é melhor, digamos, para altura de espiga, o genótipo com a média mais baixa terá \(rY_i = 100\) após o reescalonamento. O genótipo com o menor WAASB terá \(rW_i = 100\). De fato, o índice WAASBY já é computado com a função waasb(), então, agora, basta extrair-mos os valores
waasby_ind <- gmd(model_waasb, what = "WAASBY")
## Class of the model: waasb
## Variable extracted: WAASBY
print_tbl(waasby_ind, digits = 2)
| GEN | ALT_PLANT | ALT_ESP | COMPES | DIAMES | COMP_SAB | DIAM_SAB | MGE | NFIL | MMG | NGE |
|---|---|---|---|---|---|---|---|---|---|---|
| H1 | 56.11 | 50.00 | 77.57 | 66.95 | 59.29 | 75.15 | 81.33 | 71.33 | 67.43 | 37.13 |
| H10 | 15.67 | 30.34 | 70.88 | 18.77 | 12.46 | 76.95 | 53.08 | 33.31 | 36.89 | 55.36 |
| H11 | 50.49 | 35.19 | 77.99 | 44.89 | 40.60 | 82.94 | 70.55 | 15.99 | 66.65 | 51.53 |
| H12 | 32.40 | 15.51 | 10.74 | 59.58 | 32.80 | 0.00 | 27.40 | 72.03 | 42.44 | 39.86 |
| H13 | 60.66 | 51.56 | 53.71 | 54.64 | 45.26 | 54.11 | 65.77 | 50.00 | 77.44 | 45.99 |
| H2 | 55.35 | 31.89 | 73.47 | 42.27 | 25.21 | 56.32 | 56.61 | 38.98 | 55.68 | 60.34 |
| H3 | 45.68 | 39.07 | 15.64 | 69.09 | 42.06 | 45.92 | 48.36 | 31.83 | 54.28 | 27.47 |
| H4 | 64.55 | 54.09 | 75.48 | 69.57 | 46.85 | 76.07 | 73.83 | 26.27 | 57.94 | 93.69 |
| H5 | 91.30 | 72.23 | 72.79 | 67.42 | 78.05 | 80.59 | 80.66 | 30.84 | 77.51 | 76.52 |
| H6 | 71.67 | 67.13 | 50.00 | 83.57 | 73.40 | 51.37 | 53.89 | 57.77 | 96.81 | 31.23 |
| H7 | 53.15 | 54.14 | 52.44 | 73.83 | 78.55 | 48.39 | 42.71 | 63.26 | 59.24 | 46.29 |
| H8 | 3.72 | 2.32 | 38.69 | 35.11 | 16.28 | 41.53 | 15.75 | 52.27 | 16.85 | 27.26 |
| H9 | 47.17 | 61.03 | 47.35 | 5.61 | 35.04 | 58.49 | 0.00 | 27.84 | 0.00 | 17.29 |
plot_waasby(model_waasb, var = "MMG")
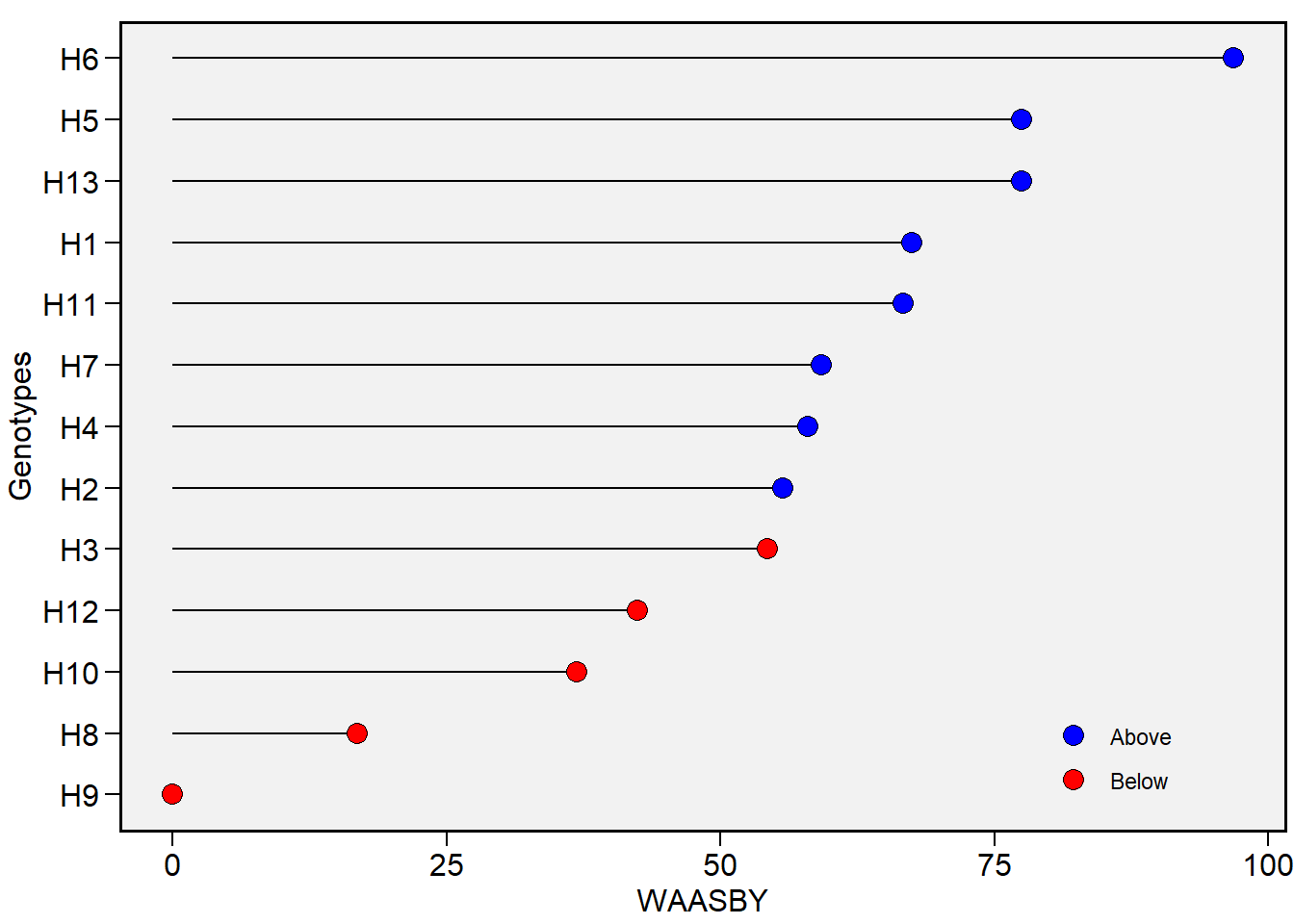
Índices multivariados
Índice de estabilidade multitrait (MTSI)
A função mtsi() é usada para calcular o índice de estabilidade multi-trait (MTSI)2. Neste caso, com o modelo calculado com os argumentos padrões, todas as variáveis analizadas são para ter ganhos positivos desejados.
mtsi_model <- mtsi(model_waasb, verbose = FALSE)
# Autovalores e variância explicada
print_tbl(mtsi_model$PCA)
| PC | Eigenvalues | Variance (%) | Cum. variance (%) |
|---|---|---|---|
| PC1 | 4.914 | 49.141 | 49.141 |
| PC2 | 2.550 | 25.502 | 74.643 |
| PC3 | 1.122 | 11.217 | 85.860 |
| PC4 | 0.613 | 6.134 | 91.994 |
| PC5 | 0.403 | 4.031 | 96.025 |
| PC6 | 0.198 | 1.984 | 98.009 |
| PC7 | 0.090 | 0.899 | 98.908 |
| PC8 | 0.067 | 0.668 | 99.575 |
| PC9 | 0.038 | 0.380 | 99.955 |
| PC10 | 0.004 | 0.045 | 100.000 |
# Diferencial de seleção para estabilidade
print_tbl(mtsi_model$sel_dif_stab)
| TRAIT | Xo | Xs | SD | SDperc |
|---|---|---|---|---|
| ALT_PLANT | 0.265 | 0.245 | -0.020 | -7.583 |
| ALT_ESP | 0.236 | 0.225 | -0.011 | -4.477 |
| COMPES | 0.339 | 0.193 | -0.146 | -43.109 |
| DIAMES | 0.598 | 0.580 | -0.018 | -3.011 |
| COMP_SAB | 0.657 | 0.584 | -0.074 | -11.202 |
| DIAM_SAB | 0.354 | 0.233 | -0.121 | -34.269 |
| MGE | 1.995 | 1.170 | -0.825 | -41.364 |
| NFIL | 0.422 | 0.411 | -0.010 | -2.446 |
| MMG | 2.440 | 1.982 | -0.458 | -18.783 |
| NGE | 2.133 | 2.348 | 0.215 | 10.091 |
# Diferencial de seleção para performance
print_tbl(mtsi_model$sel_dif_trait)
| VAR | Factor | Xo | Xs | SD | SDperc | h2 | SG | SGperc | sense | goal |
|---|---|---|---|---|---|---|---|---|---|---|
| ALT_PLANT | FA 1 | 2.485 | 2.595 | 0.110 | 4.421 | 0.035 | 0.004 | 0.155 | increase | 100 |
| ALT_ESP | FA 1 | 1.343 | 1.438 | 0.095 | 7.042 | 0.000 | 0.000 | 0.000 | increase | 100 |
| COMP_SAB | FA 1 | 29.011 | 29.715 | 0.705 | 2.429 | 0.000 | 0.000 | 0.000 | increase | 100 |
| COMPES | FA 2 | 15.163 | 15.338 | 0.175 | 1.156 | 0.000 | 0.000 | 0.000 | increase | 100 |
| DIAM_SAB | FA 2 | 15.970 | 16.150 | 0.180 | 1.128 | 0.134 | 0.024 | 0.151 | increase | 100 |
| NFIL | FA 2 | 16.123 | 16.367 | 0.244 | 1.511 | 0.000 | 0.000 | 0.000 | increase | 100 |
| DIAMES | FA 3 | 49.537 | 50.538 | 1.001 | 2.020 | 0.377 | 0.377 | 0.761 | increase | 100 |
| MGE | FA 3 | 172.939 | 183.691 | 10.752 | 6.217 | 0.204 | 2.197 | 1.270 | increase | 100 |
| MMG | FA 3 | 338.666 | 352.773 | 14.107 | 4.165 | 0.000 | 0.000 | 0.000 | increase | 100 |
| NGE | FA 3 | 511.644 | 524.125 | 12.481 | 2.439 | 0.000 | 0.000 | 0.000 | increase | 100 |
Índice MGIDI
O índice MGIDI[] pode ser visto como o índice MTSI com um peso de 100 para o desempenho médio. Este índice é calculado com a função mgidi(). Aqui, usaremos os dados de exemplo df_g.
# dados
df_g <- import("http://bit.ly/df_g", setclass = "tbl")
gen_mod <-
gamem(df_g,
gen = GEN,
rep = BLOCO,
resp = everything(),
verbose = FALSE)
mgidi_mod <- mgidi(gen_mod)
##
## -------------------------------------------------------------------------------
## Principal Component Analysis
## -------------------------------------------------------------------------------
## # A tibble: 10 x 4
## PC Eigenvalues `Variance (%)` `Cum. variance (%)`
## <chr> <dbl> <dbl> <dbl>
## 1 PC1 6.63 66.3 66.3
## 2 PC2 1.59 15.9 82.2
## 3 PC3 1.14 11.4 93.6
## 4 PC4 0.46 4.6 98.2
## 5 PC5 0.14 1.39 99.6
## 6 PC6 0.03 0.26 99.8
## 7 PC7 0.01 0.11 99.9
## 8 PC8 0.01 0.05 100.
## 9 PC9 0 0.01 100
## 10 PC10 0 0 100
## -------------------------------------------------------------------------------
## Factor Analysis - factorial loadings after rotation-
## -------------------------------------------------------------------------------
## # A tibble: 10 x 6
## VAR FA1 FA2 FA3 Communality Uniquenesses
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ALT_PLANT -0.83 -0.38 0.2 0.87 0.13
## 2 ALT_ESP -0.84 -0.29 0.23 0.85 0.15
## 3 COMPES -0.39 -0.92 -0.05 0.99 0.01
## 4 DIAMES -0.84 -0.38 0.29 0.94 0.06
## 5 COMP_SAB -0.88 0 0.18 0.8 0.2
## 6 DIAM_SAB -0.36 -0.91 -0.03 0.95 0.05
## 7 MGE -0.74 -0.65 0 0.98 0.02
## 8 NFIL -0.21 -0.08 0.97 0.99 0.01
## 9 MMG -0.93 -0.28 -0.2 0.98 0.02
## 10 NGE -0.04 -0.94 0.33 0.99 0.01
## -------------------------------------------------------------------------------
## Comunalit Mean: 0.9358222
## -------------------------------------------------------------------------------
## Selection differential
## -------------------------------------------------------------------------------
## # A tibble: 10 x 11
## VAR Factor Xo Xs SD SDperc h2 SG SGperc sense goal
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 ALT_PLA~ FA1 2.46 2.87 0.412 16.8 0.973 0.401 16.3 increa~ 100
## 2 ALT_ESP FA1 1.31 1.64 0.327 24.9 0.978 0.319 24.3 increa~ 100
## 3 DIAMES FA1 48.7 52.1 3.35 6.87 0.913 3.06 6.28 increa~ 100
## 4 COMP_SAB FA1 28.5 29.7 1.26 4.43 0.942 1.19 4.17 increa~ 100
## 5 MGE FA1 168. 214. 45.3 26.9 0.933 42.2 25.1 increa~ 100
## 6 MMG FA1 334. 378. 43.7 13.1 0.929 40.6 12.2 increa~ 100
## 7 COMPES FA2 15.2 16.4 1.15 7.53 0.831 0.953 6.26 increa~ 100
## 8 DIAM_SAB FA2 15.9 16.9 1.00 6.32 0.824 0.828 5.21 increa~ 100
## 9 NGE FA2 505. 556. 51.4 10.2 0.715 36.7 7.28 increa~ 100
## 10 NFIL FA3 15.8 16.9 1.10 6.97 0.784 0.863 5.47 increa~ 100
## ------------------------------------------------------------------------------
## Selected genotypes
## -------------------------------------------------------------------------------
## H2 H6
## -------------------------------------------------------------------------------
# radar plot
plot(mgidi_mod)

# pontos fortes e fracos
plot(mgidi_mod, type = "contribution", genotypes = "all")

print_tbl(mgidi_mod$sel_dif)
| VAR | Factor | Xo | Xs | SD | SDperc | h2 | SG | SGperc | sense | goal |
|---|---|---|---|---|---|---|---|---|---|---|
| ALT_PLANT | FA1 | 2.462 | 2.874 | 0.412 | 16.753 | 0.973 | 0.401 | 16.305 | increase | 100 |
| ALT_ESP | FA1 | 1.313 | 1.640 | 0.327 | 24.873 | 0.978 | 0.319 | 24.328 | increase | 100 |
| DIAMES | FA1 | 48.739 | 52.087 | 3.349 | 6.870 | 0.913 | 3.059 | 6.276 | increase | 100 |
| COMP_SAB | FA1 | 28.463 | 29.723 | 1.260 | 4.426 | 0.942 | 1.187 | 4.171 | increase | 100 |
| MGE | FA1 | 168.436 | 213.692 | 45.256 | 26.869 | 0.933 | 42.224 | 25.068 | increase | 100 |
| MMG | FA1 | 333.815 | 377.517 | 43.702 | 13.092 | 0.929 | 40.603 | 12.163 | increase | 100 |
| COMPES | FA2 | 15.233 | 16.380 | 1.147 | 7.530 | 0.831 | 0.953 | 6.259 | increase | 100 |
| DIAM_SAB | FA2 | 15.887 | 16.892 | 1.005 | 6.324 | 0.824 | 0.828 | 5.211 | increase | 100 |
| NGE | FA2 | 504.651 | 556.058 | 51.407 | 10.187 | 0.715 | 36.747 | 7.282 | increase | 100 |
| NFIL | FA3 | 15.795 | 16.896 | 1.101 | 6.973 | 0.784 | 0.863 | 5.466 | increase | 100 |
-
Olivoto, T., Lúcio, A. D. C., Silva, J. A. G., Marchioro, V. S., Souza, V. Q., & Jost, E. (2019). Mean Performance and Stability in Multi‐Environment Trials I: Combining Features of AMMI and BLUP Techniques. Agronomy Journal, 111(6), 2949–2960. https://doi.org/10.2134/agronj2019.03.0220 ↩︎
-
Olivoto, T., Lúcio, A. D. C., Silva, J. A. G., Sari, B. G., & Diel, M. I. (2019). Mean Performance and Stability in Multi‐Environment Trials II: Selection Based on Multiple Traits. Agronomy Journal, 111(6), 2961–2969. https://doi.org/10.2134/agronj2019.03.0221 ↩︎